
SECOND LINE OF DEFENSE
Interdisziplinärer Laborverbund
SARS-CoV-2 Diagnostik am Standort Garching
Tobias Kamholz a, Markus Hergenröther a, Judith Emmler a, Tim Böhmer a,
Rosina Ehmann c, Wolfgang Bock b, Roman Wölfel c, Thomas Zimmermann a
a Zentrales Institut des Sanitätsdienstes der Bundeswehr München
b Bundeswehrkrankenhaus Ulm Abt. XXI
c Institut für Mikrobiologie der Bundeswehr
Einleitung
Ende Januar 2020 wurden die ersten Infektionen der von China ausgehenden COVID-19-Pandemie auch in Deutschland nachgewiesen, zunächst in einem gut isolierten, firmenassoziierten Ausbruchsgeschehen im Großraum München, das auf eine chinesische Geschäftsreisende zurückgeführt werden konnte. Bereits ab Februar entwickelten sich in Deutschland weitere, unabhängige Ausbruchsherde mit komplexen Viruseintragsmustern, deren ursprüngliche Quellen nicht immer sicher ermittelt werden konnten. Die zunehmend verschärfte Dynamik der Epidemie ließ in den darauffolgenden Wochen eine lückenlose Rückverfolgung der Infektionsketten bald nicht mehr zu, sodass realistische Einschätzungen bezüglich der für Deutschland – sowie auch für die Bundeswehr – zu erwartenden Fallzahlen zunehmend schwieriger wurden.
Zur Vorbereitung auf den Pandemiefall und um die damals sowohl im zivilen Gesundheitswesen als auch im Bereich der Bundeswehr nur begrenzt verfügbaren Laborkapazitäten zur SARS-CoV-2 Diagnostik schnellstmöglich und effektiv zu erweitern, wurde auf Weisung Kommando Sanitätsdienst der Bundeswehr (Kdo SanDstBw) am Standort Garching (Christoph-Probst-Kaserne) ein interdisziplinärer Laborverbund gegründet. In dem Kooperationsprojekt zwischen der Abteilung XXI Mikrobiologie/Krankenhaushygiene des Bundeswehrkrankenhauses (BwKrhs) Ulm sowie der Abteilung A (Veterinärmedizin) des Zentralen Institutes des Sanitätsdienstes der Bundeswehr (ZInstSanBw) München wurde ein Unterstützungselement zur Diagnostik des neuartigen Coronavirus aufgestellt.
PCR-basierte Verfahren zum Nachweis des damals neu aufgetretenen Coronavirus wurden von internationalen Expertengremien bereits Tage nach Veröffentlichung der ersten Genominformationen entwickelt. Das Institut für Mikrobiologie der Bundeswehr etablierte als federführende Institution für den medizinischen B-Schutz diese Nachweisverfahren umgehend zu Diagnostik- und Forschungszwecken sowie zum Methoden- und Kompetenztransfer an andere Einrichtungen der Bundeswehr.
Zum Nachweis des erfolgreichen Methodentransfers durch das ZInstSanBw München wurde die dort ad-hoc aufgebaute Diagnostik schließlich im Schulterschluss mit den Fachärzten des BwKrhs Ulm sowie unter substanzieller Unterstützung und in engem fachlichem Austausch mit den Experten am InstMikroBioBw mithilfe bereits getesteter Patientenproben validiert.
Die neben dem Routinebetrieb etablierte Fähigkeit zur unterstützenden Diagnostik steht nunmehr seit Ende April 2020 komplementär zu den am InstMikroBioBw sowie BwKrhs Ulm verfügbaren Laborkapazitäten im Sinne einer „Second line of Defense“ auch am Standort in Garching zur Verfügung.
Material und Methoden
Nach erfolgtem Methodentransfer wurde die Untersuchungsmethode zunächst mittels Kontrollmaterial aus dem InstMikroBioBw sowie aus Trachealsekreten des BwKrhs Ulm in der Abteilung A des ZInstSanBw München erprobt und erfolgreich validiert.
Aus dem Patientenprobenmaterial (i. d. R. Regel Tupfer mit Nasopharynx- bzw. Oropharynxabstrichen) wird im ersten Schritt die potenziell vorhandene Virus-RNA unter Verwendung eines kommerziell verfügbaren Aufreinigungskits (QIAmp® Viral RNA Mini Kit der Fa. Qiagen) mittels Säulenaufreinigung extrahiert.
Die aus der Patientenprobe isolierte Virus-RNA wird anschließend in einem real-time-RT (Reverse Transkriptase)-PCR-Verfahren (Primer/Sonden-Mischung von Fa. TIB MOLBIOL) auf zwei möglichen Geräteplattformen (LC96 der Fa. Roche bzw. CFX96 der Fa. BioRad) nach dem Protokoll von CORMAN et. al. (2020) vervielfältigt und dadurch nachgewiesen. Durch die gleichzeitige Validierung der SARS-CoV-2-Diagnostik an mehreren PCR-Cyclern wurde in Zeiten des weltweiten Bedarfs mit materiellen Engpässen die Flexibilität erhöht und die Durchhaltefähigkeit in der Diagnostikkapazität wirkungsvoll verbessert.
Die meisten Nachweisverfahren für SARS-CoV-2 basieren auf einem zweistufigen System mit dem Nachweis von zwei Zielsequenzen, einem sensitiveren, als Suchtest geeigneten Target sowie einem spezifischeren Target zur zweifelsfreien Bestätigung. Die häufigsten diagnostisch eingesetzten Zielgene von SARS-CoV-2 sind: N (nucleocapsid), E (envelope), S (spike) und RdRP (RNA-dependent RNA polymerase). In der aktuell am ZInstSanBw München eingesetzten Methode wird mit dem Nachweis des E-Gens sehr sensitiv auf SARS-CoV-2 und eng verwandte Viren gescreent. Ein positives Ergebnis wird anschließend durch den Nachweis eines für SARS-CoV-2 spezifischen Abschnitts im RdRP-Gen bestätigt.
Auch die am ZInstSanBw München in der Folge etablierte und auf die Geräte der Abteilung Veterinärmedizin adaptierte Multiplex-Methode weist neben einer internen Kontrolle zwei Zielsequenzen gemäß Protokoll der Fa. TIB MOLBIOL (E-Gen, RdRP-Gen) in einem einzigen Reaktionsansatz nach, sodass im Fall eines erhöhten Probenaufkommens Ressourcen geschont werden können. Hierfür wurde zur Etablierung eine zusätzliche fluoreszenzmarkierte Primer/Sonden-Kombination zum Nachweis des E-Gens angewendet:
Tab. 1: RNA-Sequenzen zum Nachweis des E-Gens bei SARS-CoV-2

Ergebnisse
Die bis Ende Juni 2020 durch den Laborverbund untersuchten Patientenproben entstammen den regionalen Sanitätseinrichtungen im Einzugsbereich der Sanitätsunterstützungszentren (SanUstgZ) Hammelburg, Kümmersbruck, München und Stetten a. k. M. Zusätzlich wurden auch Patientenproben aus dem BwKrhs Ulm im Rahmen der Methodenvalidierung untersucht.
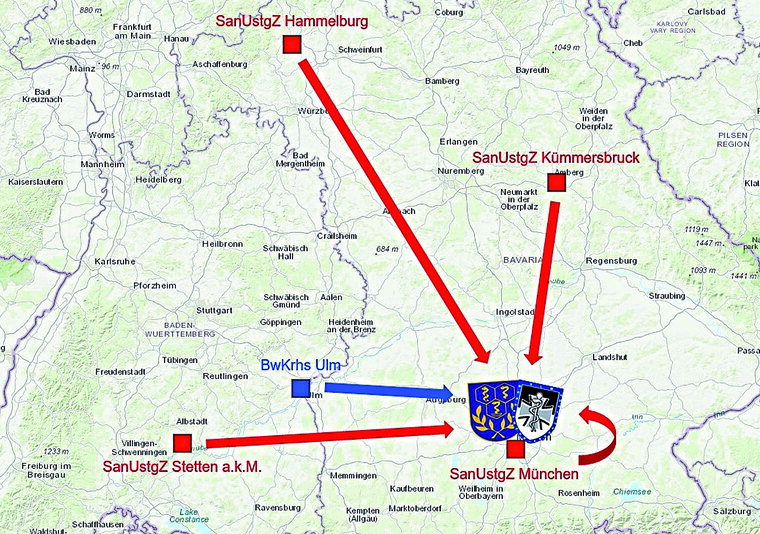
Abb. 1: Einzugsbereich für den SARS-CoV-2-Nachweis am ZInstSanBw München
In diesem fach- sowie dienstellenübergreifenden Projekt wirken mehrere Approbationen bzw. Fachrichtungen synergistisch zusammen:
- Auftragsverwaltung und Befundung erfolgen durch das fachärztliche Personal der Abt. XXI (Mikrobiologie/Krankenhaushygiene) des BwKrhs Ulm.
- Die labormedizinischen Untersuchungen werden durch das Laborfachpersonal der Abt A (Veterinärmedizin) des ZInstSanBw München durchgeführt.
- Die Gesamtkoordination des Projektes wird durch die Abt. F (Medizinischer ABC-Schutz) der Sanitätsakademie der Bundeswehr im Auftrag von Kdo SanDstBw VII-2 (Einsatzführungszentrum) sichergestellt.
Diskussion/Fazit
Die ambulante truppenärztliche Versorgung in Bayern und Baden-Württemberg konnte durch die zusätzliche Etablierung nutzbarer Laborkapazitäten aus dem Bereich der Routine innerhalb kürzester Zeit wirkungsvoll verstärkt und damit die am InstMikroBioBw sowie BwKrhs Ulm vorhandenen fachärztlichen Ressourcen entlastet werden.
Am Standort Garching ist damit ein Modell für einen interdisziplinären Laborverbund zwischen Veterinärmedizinern und Humanmedizinern entstanden, welches es ermöglicht, auch einen in Zukunft möglicherweise steigenden Proben- und Beprobungszahlen in Bayern und Baden-Württemberg zu begegnen. Für eine mögliche zweite Infektionswelle können so schnell zusätzliche Laborkapazitäten freigesetzt bzw. zur Entlastung anderweitig gebundener Dienststellen aufgebaut und damit fachliche Kräfte und Ressourcen gebündelt werden.
Nach dem Motto „Gemeinsam stark durch die Krise“ könnten solche Zusammenschlüsse und Synergien auch als ein weiteres wegweisendes Modell für die zukünftige Bewältigung ähnlicher Krisenszenarien dienen.
Literatur
- Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz, Anwendungsempfehlungen für den Nachweis von Antikörpern bei SARS-CoV-2 vom 14. Mai 2020; https://www.wko.at/branchen/t/handel/200514_Anwendungsempfehlungen-fuer-den-Nachweis-von-Antikoer.pdf, letzter Aufruf 17. August 2020.
- Corman, V. M. et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT–PCR. Euro Surveill 2020; 25(3): 2000045.
- Drosten C, Chiu LL, Paning M et al.: Evaluation of advanced reverse transcription–PCR assays and an alternative PCR target region for detection of severe acute respiratory syndrome-associated coronavirus. J Clin Microbiol. 2002; 42(5): 2043-2047.
- Guber F, Hufnagl P: Vergleich unterschiedlicher Nukleinsäure-Amplifikationssysteme für SARS-CoV-2. ingenetix 2020; https://www.ingenetix.com/vergleich-unterschiedlicher-nukleinsaeure-amplifikationssysteme-fuer-sars-cov-2/, letzter Aufruf 17. August 2020.
- Robert Koch Institut: COVID-19 (Coronavirus SARS-Cov-2). RKI 2020; https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/nCoV.html, letzter Aufruf 17. August 2020.
- Wölfel R, Corman VM, Guggemos W et al.: Virological assessment of hospitalized patients with COVID-2019. Nature 2020; 581: 465–469.
Für die Verfasser
Oberstveterinär Dr. Judith Emmler
Zentrales Institut des Sanitätsdienstes der Bundeswehr München
Ingolstädter Landstraße 102, 85748 Garching
E-Mail: JudithEmmler@bundeswehr.org