Etablierung einer robusten SARS-CoV-2-Back-Up-Diagnostik und deren Anwendung zur Evaluierung von Probenahme und -bearbeitung für die Human- und Veterinärmedizin (Vortrags-Abstract)
Anja Petrova
a Zentrales Institut des Sanitätsdienstes der Bundeswehr, Kiel
Hintergrund
Die rasante Ausbreitung des neuartigen Coronavirus SARS-CoV-2 auf globaler Ebene erforderte die Etablierung einer zuverlässigen molekularbiologischen Hochdurchsatz-Diagnostik für ein bis dato unbekanntes Virusisolat innerhalb kürzester Zeit. Dies stellte sowohl diagnostische Laboratorien der Bundeswehr als auch Hersteller von notwendigen Laborreagenzien vor eine immense Herausforderung. Das Resultat war eine Verzögerung der flächendeckenden Anwendung der SARS-CoV-2-Diagnostik sowie Lieferengpässe von RT-PCR-Reagenzien.
Zur Gewährleistung einer schnellstmöglichen Patientenversorgung etablierten Krankenhäuser und Labore ihre Diagnostik weitestgehend mit gebrauchsfertigen SARS-CoV-2-RT-PCR-Kits, welche jedoch nur limitiert verfügbar waren und außerdem intransparent hinsichtlich Art und Zuverlässigkeit des SARS-CoV-2-Nachweises. Personelle und zeitliche Engpässe in Krankenhäusern ließen jedoch oftmals nicht die Etablierung einer eigenen (inhouse) Methode zu.
Im Sinne des One-Health-Gedanken wurde das Bundeswehrkrankenhaus Berlin von der veterinärmedizinischen Abteilung des Zentralen Instituts des Sanitätsdienstes der Bundeswehr Kiel (ZInstSanBw Kiel) unterstützt. Das Institut ist keine Ressortforschungseinrichtung, sondern erfüllt überwiegend umfangreiche Diagnostikaufgaben.
Die Unterstützungsleistung erfolgte in methodischer Hinsicht durch Entwicklung einer robusten SARS-CoV-2-Back-Up-RT-PCR mit erhöhter Herstellerunabhängigkeit (erste Fragestellung). Zur Gewährleistung der akkuraten SARS-CoV-2-Diagnostik bei Soldatinnen und Soldaten wurde diese Methode nachfolgend zur Evaluierung von erfolgsentscheidenden vorgeschalteten Schritten in der Diagnostikkette angewandt (zweite Fragestellung). Darüber hinaus erforderte das zoonotische Potential des Erregers – und somit eine potenzielle Gefährdung von Diensthundeführern – sowie die Einführung einer Meldepflicht bei Haustieren die Verifizierung dieser Methode für die Veterinärmedizin mit Fokus auf das Diensthundewesen der Bundeswehr (dritte Fragestellung).
Materialien und Methoden
Entwicklung eines Back-Up real-time RT-PCR Verfahrens
Basierend auf 4 von der WHO gelisteten Methoden (E-Gen/Corman et al., 2020; N-Gen/CDC, USA, 2020 als Screening; RdRP-Gen/Institut Pasteur Frankreich und Corman et al., 2020 als Bestätigung) wurden zunächst Einzelassays mit dem CFX96 Real-Time-System (BioRad®) mit insgesamt 5 offenen RT-PCR-Chemien analysiert. Die besten Assays (E-Gen/Corman et al., 2020; RdRP-Gen/Institut Pasteur Frankreich) wurden zu Duplex-Verfahren erweitert; hier erfolgte im ersten Schritt der Nachweis einer Internal-Control (IC)-RNA (Indical) mit EGFP-Mix 1 (Hoffmann et al. 2006) und im zweiten Schritt der β-Actin-Nachweis (Wernike et al. 2011).
Dies erfolgte mit vier offenen RT-PCR-Chemien (u. a. Luna Universal One-Step RT-qPCR-Kit (NEB) und AgPath-ID One-Step RT-PCR-Reagents (Thermo Fisher Scientific)) mit einem reduzierten Gesamtreaktionsvolumen. Sensitivität und Spezifität wurden anhand von humanen und ovinen Oraltupfern überprüft. Die Test-Performance wurde u. a. durch Vergleich mit 7 zertifizierten SARS-CoV-2-PCR-Kits verifiziert. Die Testung erfolgte weiterhin mit einer für den Auslandseinsatz bereitgestellten PCR-Plattform (Smart-Cycler) und lyophilisierter PCR-Chemie.
Zusätzlich wurde eine RT-PCR zur Identifizierung aller Vertreter der β-Coronaviren eingefahren.
Evaluierung vorgeschalteter Parameter
Diese Parameter umfassten (Abbildung 1):
- Einfluss von speziesspezifischen Sputum auf die inhouse RT-PCR,
- Robustheit von SARS-CoV-2 in Oraltupfern (= nicht aufgereinigtes Probenmaterial),
- Robustheit von SARS-CoV-2-RNA (= aufgereinigtes Probenmaterial) sowie
- Effekte einer Hitzevorbehandlung.
Drei Oraltupfer (eSwabsTM (Copan), Xpert Nasal (Cepheid®), Sigma Virocult® (MWE)) wurden zur Analyse des Einflusses von speziesspezifischem Sputum nativ und nach oraler Anwendung bei Mensch und Hund mit unterschiedlichen SARS-CoV-2-Konzentrationen versetzt (Spike) und in der RT-PCR getestet.
Zur Analyse der Robustheit von SARS-CoV-2 in Oraltupfern wurden humane und canine Oraltupfer an den Tagen 0, 7, 14 und 21 post Probenahme/Spike nach Lagerung bei 4 °C sowie bei Raumtemperatur jeweils mit und ohne Hitzevorbehandlung (30 min, 56 °C) extrahiert und mittels RT-PCR untersucht.
Die Robustheit von SARS-CoV-2-RNA aus Zellkulturüberstand wurde mit verschiedenen Extraktionsreagenzien, Lagertemperaturen/-zeiten sowie Gefrier-Tau-Zyklen analysiert. Mit Humantupfer-RNA wurde ein Langzeitlagertest über 13 Wochen bei 4 °C durchgeführt. Auch die Lagerfähigkeit von Hundetupfer-RNA wurde untersucht.
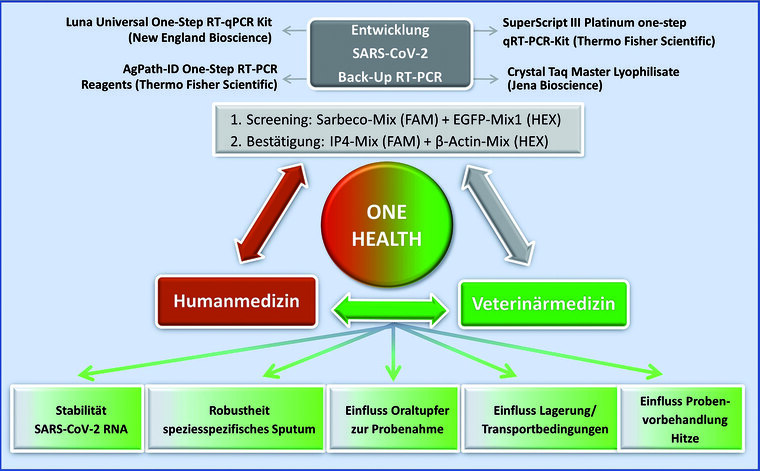
Abb. 1: Darstellung der komplexen Entwicklungsarbeit zur Sicherung der SARS-CoV-2-Diagnostik in Human- und Veterinärmedizin
Ergebnisse
Zuerst wurde ein zweistufiges inhouse TaqMan®-basiertes real-time RT-PCR-Assay etabliert, bestehend aus einem ersten Screening zum Nachweis verschiedener β-Coronaviren des Subgenus Sarbeco und einer nachfolgenden Bestätigung von COVID-19, jeweils mit simultanem Nachweis einer internen Kontrolle (Duplex-Verfahren). Dieses Assay erwies sich als kompatibel mit vier offenen RT-PCR-Chemien und wurde außerdem mit einer für den Auslandseinsatz bereitgestellten PCR-Plattform verifiziert. Ergänzend wurde ein RT-PCR-Verfahren zur Detektion aller β-Coronaviren etabliert.
Nachfolgend wurde gezeigt, dass weder humanes noch canines Sputum das inhouse Assay beeinflusst. Die Analyse der Robustheit von SARS-CoV-2 in Oraltupfern in Form von Lagerungsversuchen erbrachte bei Mensch und Hund ähnliche RT-PCR-Ergebnisse (Abbildung 2). Die größten Verluste waren stets innerhalb der ersten Woche vorhanden. Während bei Cepheid® und MWE nachfolgend ein weitgehend konstanter SARS-CoV-2-Genomnachweis möglich war, zeigten eSwabs™ deutliche Verluste mit überwiegend negativen Ergebnissen nach drei Wochen Lagerung bei Raumtemperatur.
Die Probenvorbehandlung mit Hitze (56 °C, 30 min) wirkte sich in der Mehrzahl negativ aus (siehe Tabelle 1), vor allem bei eSwabs™.
Die SARS-CoV-2-RNA aus Zellkulturüberstand zeigte keine Verluste nach Lagerung bei 4, -20 und -80 °C über zwei Wochen sowie nach drei Gefrier-Tau-Zyklen. Ein RNA-Langzeitlagertest aus humanen Oraltupfern über 13 Wochen bei 4 °C ergab nur geringfügige Ct-Wert-Verluste (< 1 Log-Stufe) mit lediglich einem qualitativen Verlust bei insgesamt 23 Proben. Im Gegensatz dazu wurden bei Hundetupfer-Eluaten bereits nach Über-Nacht-Lagerung geringfügige Ct-Wert-Verluste detektiert.
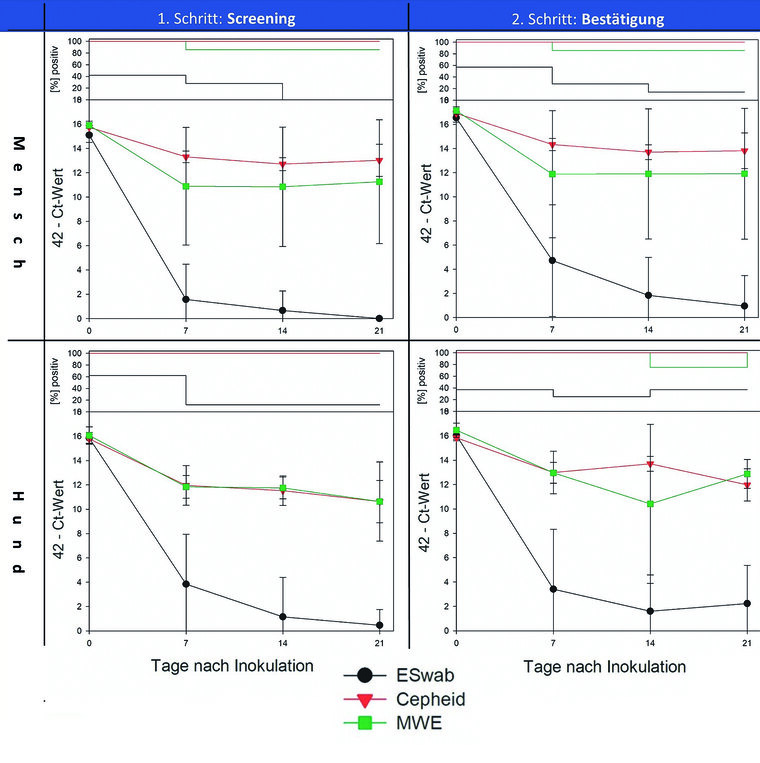
Abb. 2: SARS-CoV-2-Genomnachweise aus einem dreiwöchigen Lagerungstest bei Raumtemperatur: Evaluiert wurden drei Oraltupfersorten, welche jeweils bei Mensch und Hund angewendet wurden. In den jeweils zweiteiligen Diagrammen illustriert der obere Teil den prozentualen Anteil an SARS-CoV-2-positiven Tupferproben zum jeweiligen Zeitpunkt, der untere Teil zeigt das jeweilige RT-PCR-Ergebnis.
Tab. 1: Effekte der Hitzevorbehandlung auf die RT-PCR-Ergebnisse von 3 Oraltupfersorten, angewendet bei Mensch und Hund und Lagerung über 3 Wochen bei Raumtemperatur (RT)
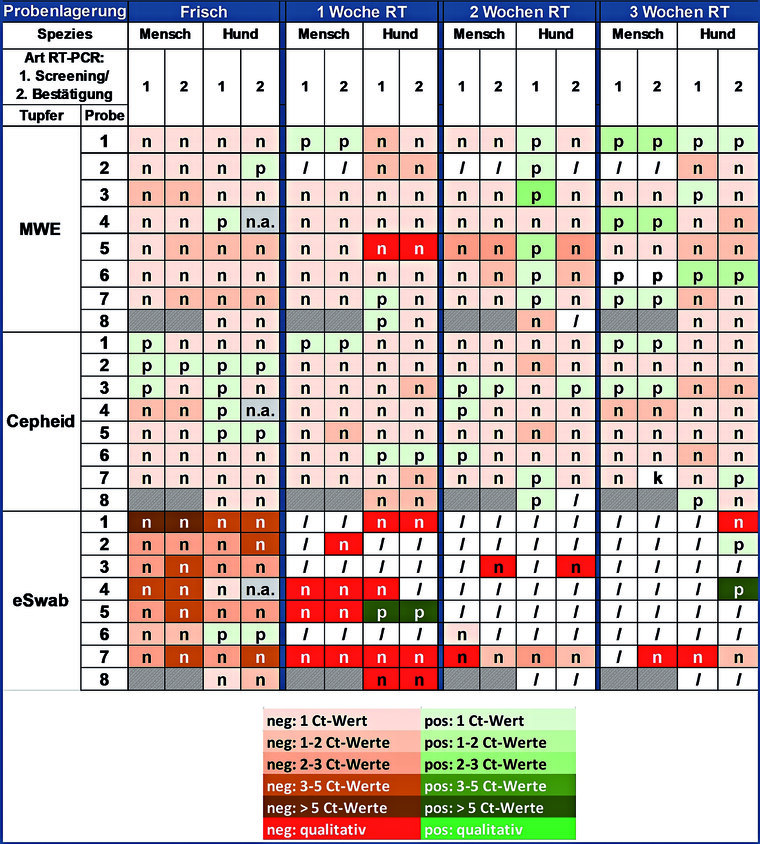
n = negativer Effekt; p = positiver Effekt; / = Probe bereits zum vorherigen Zeitpunkt negativ getestet; k = keine Änderung durch Hitze; n.a. = nicht auswertbar
Diskussion
Die als Unterstützungsleistung für das Bundeswehrkrankenhaus Berlin etablierte Back-Up-SARS-CoV-2-RT-PCR bietet gegenüber kommerziellen Testkits folgende Vorteile:
- Die größere Herstellerunabhängigkeit erhöht das Ausmaß an Flexibilität, was für Krisenvorsorge und Engpassmanagement von hohem Wert ist.
- Die volle Transparenz ermöglicht eine rasche Reaktion auf Mutationen.
- Das reduzierte Gesamtreaktionsvolumen führt zur signifikanten Kosteneinsparung und ermöglicht eine SARS-CoV-2-Diagnostik bei Soldatinnen und Soldaten auch unter Bedingungen limitierter Ressourcen.
- Die Angleichung der PCR-Konditionen beider Testschritte ermöglicht eine schnelle Identifizierung und somit auch Separierung von Virusträgern.
- Die Methode wurde auch für ein Einsatz-PCR-System verifiziert; sie ist einsatztauglich.
Durch die zusätzliche Etablierung einer „Breitspektrum“-RT-PCR-Methode für β-Coronaviren wurde der diagnostische Umfang erweitert und die Möglichkeit zur Differenzierung des SARS-CoV-2 gegen andere Virusvertreter geschaffen, deren Vorkommen v. a. im Auslandseinsatz denkbar ist.
Die SARS-CoV-2-inhouse-RT-PCR erwies sich als gleichermaßen geeignet für Mensch und Hund. Die drei analysierten Oraltupfer befinden sich in Bundeswehrkrankenhäusern (eSwabs™ und Cepheid®) und Diagnostiklaboren (MWE) in Anwendung, wobei bei deren Auswahl die limitierte Marktverfügbarkeit eine große Rolle spielt. Die Studie zeigt eine deutlich schlechtere Eignung von eSwabs™ im Vergleich zu MWE- und Cepheid®-Tupfern (Abbildung 2). Dies wirkt sich vor allem dann fatal auf die Diagnostik aus, wenn die Probenaufreinigung nicht sofort nach der Probenahme erfolgt, z. B. bei Probentransport aus Gruppenunterkünften ins Diagnostiklabor oder bei Probenlagerung im Krankenhaus bis zur Aufreinigung. Zudem weisen Ergebnisse aus Pilotversuchen auf die Dringlichkeit einer schnellstmöglichen Kühlung von nicht-aufgereinigtem Probenmaterial hin.
Vor allem aber ist eine schnelle Nukleinsäureextraktion nach Probenahme von essenzieller Bedeutung. SARS-CoV-2-RNA aus Zellkulturüberstand und humanen Proben hat sich als sehr robust erwiesen. Im Gegensatz dazu scheint SARS-CoV-2-RNA aus Hundetupfern empfindlicher gegenüber Kühlschranklagerung zu sein, weshalb hier eine schnelle Tiefkühlung entscheidend ist.
Eine Probenvorbehandlung durch Hitze wird oftmals in der veterinärmedizinischen Diagnostik zur Inaktivierung von Inhibitoren in potenziell verunreinigten Probenmaterialien durchgeführt, z. B. bei der Tierseuchendiagnostik von Wildtieren auf Truppenübungsplätzen der Bundeswehr. Aufgrund der mehrheitlich negativen Effekte (v. a. bei eSwabs™) ist bei der SARS-CoV-2-Diagnostik eine Hitzevorbehandlung nicht zielführend.
Fazit
Die Back-Up inhouse SARS-CoV-2 RT-PCR steht für den Einsatz in Bundeswehrkrankenhäusern, für den Auslandseinsatz sowie für das Diensthundewesen der Bundeswehr bereit.
Der Einsatz geeigneter Tupfersysteme zur Probenahme sowie eine gute Organisation von nachfolgender Probenlagerung und -transport bis zu deren Aufreinigung ist essenziell für die zuverlässige Identifizierung von Virusträgern. Mit der Methode kann ein wesentlicher Beitrag zur Verhinderung der SARS-CoV-2-Ausbreitung unter unseren Soldaten und somit zur Sicherstellung der Einsatzfähigkeit geleistet werden.
Manuskriptdaten
Zitierweise
Petrov A: Etablierung einer robusten SARS-CoV-2-Back-Up-Diagnostik und deren Anwendung zur Evaluierung von Probenahme und -bearbeitung für die Human- und Veterinärmedizin (Vortrags-Abstract). WMM 2020; 64(12): 33-36.
Verfasserin
Oberstabsveterinär Dr. Anja Petrov
Zentrales Institut des Sanitätsdienstes der Bundeswehr Kiel
Tierseuchen- und Zoonosendiagnostik
Kopperpahler Allee 120, 24119 Kronshagen
E-Mail: anjapetrov@bundeswehr.org
Vortrag beim Wettbewerb um den Heinz-Gerngroß-Förderpreis der Deutschen Gesellschaft für Wehrmedizin und Wehrpharmazie e. V. am 23. Oktober 2020 in Rostock-Warnemünde.