
ZINSTSANBW RELOADED
Gemeinsam von Null auf SARS-CoV-2-PCR
in drei Wochen – ein Erfahrungsbericht
Werner Wenzela, Anja Petrovb, Miriam Haarmannb, Daniel Bartosikc, Martin Müllerd
a Bundeswehrkrankenhaus Hamburg, Abteilung XXI – Mikrobiologie und Krankenhaushygiene
b Zentrales Institut des Sanitätsdienstes der Bundeswehr Kiel
c Universität Greifswald, Institut für Pharmazie, Abteilung Pharmazeutische Biotechnologie
d Bundeswehrkrankenhaus Berlin, Abteilung XXI – Mikrobiologie und Krankenhaushygiene
Hintergrund
Am 11. März 2020 erklärte der Generaldirektor der Weltgesundheitsorganisation (WHO) den Ausbruch von COVID-19 zur Pandemie. Spätestens zu diesem Zeitpunkt wurde deutlich, dass auch im Sanitätsdienst der Bundeswehr Vorbereitungen zu treffen waren – nicht nur, um für die Behandlung der Patienten gerüstet zu sein, sondern vor allem auch, um Diagnostik zur Unterstützung der medizinischen Versorgung bereitzuhalten.
Zu diesem Zeitpunkt waren entsprechende diagnostische Kapazitäten innerhalb der Bundeswehr lediglich an zwei Spezialeinrichtungen des Sanitätsdienstes etabliert. Das Institut für Mikrobiologie der Bundeswehr (InstMikroBioBw) in München verfügte über umfangreiche Erfahrungen mit der von Prof. Dr. Drosten, Berlin, publizierten molekularbiologischen Methode zum Nachweis von SARS-CoV-2. Auch in der Abteilung XXI –Mikrobiologie, Krankenhaushygiene – des Bundeswehrzentralkrankenhauses (BwZKrhs) Koblenz, die in der Sektion XXI B entsprechende Fähigkeiten bereithält, war das Verfahren bereits etabliert. Am Bundeswehrkrankenhaus (BwKrhs) Berlin hingegen sah die Situation anders aus, hier standen weder Personal noch Material für die dringend erforderliche Diagnostik zur Verfügung.
PCR-Diagnostik – nicht nur für die Forschung
Im Jahr 2018 wurden die Abteilungen I – Medizin – der Zentralen Institute des Sanitätsdienstes der Bundeswehr (ZInstSanBw) als neue Abteilungen XXI –Mikrobiologie, Krankenhaushygiene – an die Bw(Z)Krhs angegliedert. Die Abteilungen I der ZInstSanBw hatten bis dahin ein sehr breites medizinisch-diagnostisches Portfolio angeboten, vom Schwerpunkt Mikrobiologie über immunologische Untersuchungen bis hin zur Diagnostik von Autoimmunerkrankungen und Allergien. Auch für die Durchführung molekularbiologischer Untersuchungen – und damit den direkten Nachweis viraler Krankheitserreger – waren ausreichend Kapazitäten vorhanden. Im Zuge der Planungen zur Ausgestaltung der „Zielstruktur 2020“ wurde entschieden, dass in den neu aufgestellten klinisch-mikrobiologisch orientierten Abteilungen XXI – jenseits der klassischen Bakteriologie – Analysen in der Infektionsserologie und Molekularbiologie nur mit vollautomatisierten Systemen bzw. Fertig-Kartuschen angeboten werden. Die Fähigkeit zur Durchführung herstellerunabhängiger „offener“ Methoden wurde damit für die Abteilungen XXI in den BwKrhs Berlin, Hamburg und Ulm/München aufgegeben.
Die SARS-CoV-2-Pandemie machte es jedoch sehr kurzfristig erforderlich, genau diese offenen molekularbiologischen Methoden nicht ausschließlich als hoch spezialisierte Instrumente für epidemiologische Studien und Forschungszwecke, sondern in der Breite als notwendigen Bestandteil der Routinediagnostik verfügbar zu halten.
In zivilen Einsendelaboren dauerte die Diagnostik zu diesem Zeitpunkt in Berlin bis zu 5 Tage. Am 12. März 2020 meldete der Klinische Direktor Abteilung XXI BwKrhs Berlin die Fähigkeitslücke an Kommando Sanitätsdienst der Bundeswehr (Kdo SanDstBw). Hieraus resultierte am 13. März 2020 der Auftrag – salopp formuliert: „möglich machen, was irgendwie möglich ist“.
Mit Kooperationen zum Erfolg
Da in der Abteilung XXI des BwKrhs Berlin keine Sanitätsstabsoffiziere Arzt mehr vorhanden waren, die Erfahrung mit der Etablierung offener molekularbiologischer Methoden hatten, erging ein Ersuchen um Unterstützung an das BwKrhs Hamburg, wo der Erstautor dieses Beitrags als ehemaliger Mitarbeiter der Abteilung I ZInstSanBw Kiel, Außenstelle (ASt) Berlin, inzwischen tätig war. Der Auftrag war so einfach wie umfassend: Es sollte schnellstmöglich die Etablierung einer SARS-CoV-2-PCR am BwKrhs Berlin erfolgen.
Die Durchführung einer Real-time-PCR-Diagnostik 1 erfordert erfahrenes Personal und eine entsprechende materielle Ausstattung (siehe Tabelle 1)
Tab. 1: Überprüfung der materiellen und personellen Voraussetzungen
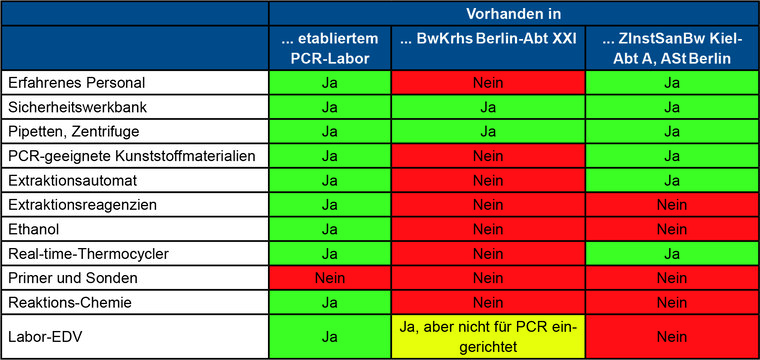
Tabelle 1 zeigt, dass in einem etablierten PCR-Labor die nötige Grundausstattung bereits vorhanden ist und nur die für den jeweils gesuchten Erreger spezifischen Primer und Sonden gekauft werden müssen. Diese sind nach Publikation des Erregergenoms meist schnell und in ausreichender Menge verfügbar, weil sie einfach zu synthetisieren sind. In der Abteilung XXI des BwKrhs Berlin war nach Aufgabe des Bereichs PCR-Diagnostik im Zuge der Umgliederung 2018 jedoch kaum noch materielle Ausstattung vorhanden. Die Aufgabe der ersten Woche war also neben einer Einweisung in die am BwZKrhs Koblenz etablierte Methodik vor allem das Beschaffen der nötigen Ausrüstung und Reagenzien.
Hilfe aus der Veterinärmedizin
Im Gegensatz zur ehemaligen Abteilung I ZInstSanBw Kiel (bis 2017) hatte die neue Abteilung A (Veterinärmedizin) ZInstSanBw Kiel in der Außenstelle Berlin mit der Umstrukturierung 2018 einen Aufwuchs erfahren. Während bis dahin nur Trinkwasseruntersuchungen durchgeführt wurden, sollen nun auch Lebensmittel getestet werden. Die hierfür erforderliche Ausstattung war 2019 teilweise zugeflossen. Ein vollständig etabliertes PCR-Labor war hier zwar ebenfalls noch nicht in Betrieb, jedoch war eine Grundausstattung vorhanden und die Laborleitung hatte Erfahrung in der PCR-Diagnostik. Im „Mutterhaus“ des ZInSanBw Kiel in Kronshagen ist eine sehr umfangreiche Kompetenz in der PCR-Diagnostik vorhanden, da hier ein Großteil der Tierseuchendiagnostik der Bundeswehr stattfindet. Es war daher naheliegend, die „alten Kontakte“ auf persönlicher und wissenschaftlicher Ebene aus den Zeiten der Zusammenarbeit am ZInstSanBw Kiel wieder aufleben zu lassen und die SARS-CoV-2-PCR-Diagnostik gemeinsam aufzubauen. So wurde die approbationsübergreifende Funktionalität des früheren ZInstSanBw Kiel für die Herausforderung SARS-CoV-2-Diagnostik wieder hergestellt – eine Situation, die als praktisches Beispiel für die enge Verflechtung von medizinischer und veterinärmedizinischer Kompetenz i.S. des „One Health“-Ansatzes dienen kann.
Problemfeld Marktverfügbarkeit
Der erste Schritt der PCR-Diagnostik ist die Extraktion der DNA/RNA. Hierfür existieren verschiedene automatisierte und manuelle Methoden. Die diesbezügliche Marktsichtung zeigte ein Problem, das das Projekt die gesamte Anfangszeit über begleiten sollte: Der Markt war „leergefegt“. Geräte zur automatisierten Extraktion hatten zu diesem Zeitpunkt bereits Lieferzeiten von mehreren Monaten. Es wurde daher beschlossen, ein in der Abteilung A ZInstSanBw Kiel am Standort Kronshagen vorhandenes Gerät zur Extraktion von 96 Proben pro Lauf nach Berlin zu verlagern. Reagenzien hierfür waren jedoch nur für einige hundert Proben vorhanden, die Herstellerfirma konnte zur Lieferbarkeit keine Aussagen treffen. Zwar stand auch in der Außenstelle ein Extraktionsautomat zur Verfügung, allerdings nur zur gleichzeitigen Bearbeitung von 14 Proben. Daher wurden zusätzlich von mehreren Herstellern Reagenzien-Kits zur manuellen Extraktion bestellt in der Hoffnung, dass zumindest ein Teil tatsächlich geliefert wird.
Flexibles Handeln gefragt
Der gesamte Prozess der Beschaffung von Verbrauchsmaterial wurde durch das Personal der Bundeswehrkrankenhausapotheke Berlin in hervorragender Weise unterstützt und gemanagt. Jede Bestellung wurde zügig bearbeitet, auch bei Lieferanten, die nicht üblicherweise beauftragt werden, und es wurde auch nach alternativen Versorgungsmöglichkeiten recherchiert. Denn neben den eigentlichen Extraktionsreagenzien ist noch an sich simples Zubehör – wie PCR-geeignete Pipettenspitzen und vor allem 96 %iger Alkohol – erforderlich. Aber letzterer war, genau wie Desinfektionsmittel, zu der Zeit am Markt so gut wie nicht erhältlich. Hier wurde aus vorhandenen Eigenbeständen durch ZInstSanBw Kiel, durch die BwKrhs-Apotheke Berlin und auch durch Abt XXI BwZKrhs Koblenz unterstützt – sonst wären das Projekt möglicherweise wegen des Fehlens von simplem Alkohol gescheitert.
Alternative Methode etabliert
Im Hinblick auf die Reagenzien für die eigentliche PCR-Reaktion wäre es naheliegend gewesen, genau die Methode anzuwenden, die durch InstMikroBioBw am BwZKrhs Koblenz bereits etabliert war. Hiervon haben wir jedoch aus mehreren Gründen Abstand genommen: Erstens waren die dafür benötigten PCR-Reagenzien ebenfalls kaum noch erhältlich, weil es die Methode war, die von den meisten Laboren durchgeführt wurde. Zweitens erfordert diese Methodik viel Erfahrung in der PCR-Diagnostik, weil die Reagenzien-Ansätze von Hand berechnet und pipettiert werden müssen. Es war jedoch absehbar, dass die Methodik in Berlin von eher wenig erfahrenem Personal durchgeführt werden würde. Drittens sollten wegen der fehlenden methodischen Erfahrungen möglichst viele Kontrollen mitgeführt werden, insbesondere eine zusätzliche Reaktion zum Nachweis des enthaltenen Untersuchungsmaterials (also zum Nachweis von menschlichem Genom). Glücklicherweise kamen zu diesem Zeitpunkt gerade etliche Hersteller mit kompletten Reagenzien-Kits auf den Markt. Von den tatsächlich (zunächst in geringen Mengen) lieferbaren Kits wurden zwei geeignete ausgewählt und bestellt.
Erste Testläufe
Im Laufe der zweiten Woche waren alle Materialien aus verschiedenen Quellen so weit vorhanden, dass am Ende der Woche mit den Testläufen begonnen werden konnte. Es wurden insgesamt 5 verschiedene Extraktionsmethoden (vier automatisierte sowie eine manuelle) unter Anwendung der 2 PCR-Methoden verglichen. Zu diesem Zeitpunkt wurden in ausreichender Zahl Patienten im BwKrhs Berlin behandelt, so dass genügend positiv charakterisierte Proben zur Testung der Methoden zur Verfügung standen. Da zudem neben der üblichen Prozesskontrolle mit zugegebener RNA auch eine Kontrolle für menschliches Genom mitgeführt wurde, konnte durch ausführliche Validierungsläufe sehr bald festgestellt werden, dass die Methodik zuverlässig funktionierte.
Herstellerunabhängigkeit angestrebt
Ergänzend zur SARS-CoV-2-Diagnostik mittels kommerzieller Real-time-PCR-Kits wurde am ZInstSanBw Kiel in Kronshagen in der Teileinheit Tierseuchen- und Zoonosen-Diagnostik eine Inhouse-Methode als Ergänzungs- und Back-up-Diagnostik von Oberstabsveterinär Dr. Petrov unter der Leitung von Oberfeldveterinär Dr. Schotte etabliert. Diese Unterstützung des humanmedizinischen Sektors seitens der veterinärmedizinischen Abteilung in Kronshagen für das BwKrhs Berlin bot sich auch in Hinblick auf die sehr ähnliche Laborausstattung für RNA-Extraktion und PCR an. Dieses Inhouse-RT-PCR-Verfahren sollte im Vergleich zur ausschließlichen Verwendung von SARS-CoV-2-spezifischen kommerziellen Testkits mehr Herstellerunabhängigkeit ermöglichen, um auch bei internationalen Lieferengpässen die Diagnostik sicherzustellen. Zudem wurde zu dieser Zeit mit sehr hohen Probenzahlen kalkuliert, so dass die preiswertere Inhouse-Methode zu einer erheblichen Kosteneinsparung geführt hätte. Weiterhin besitzt dieser Assay angesichts seiner vollen Transparenz das wertvolle Potenzial einer schnellen und flexiblen Anpassung im Erregernachweis. Falls die fortschreitende virale Evolution im Zuge der weltweiten SARS-CoV-2-Ausbreitung eine Änderung des nachzuweisenden Genomabschnittes erforderlich machen sollte, ist dies mit der Inhouse-Methode innerhalb kurzer Zeit möglich.
Im Zuge der Validierung wurde, basierend auf WHO-gelisteten Methoden, auf welche auch das Robert Koch-Institut Bezug genommen hatte, nach intensivem Austausch mit virologischen und molekularbiologischen Expertengruppen innerhalb und außerhalb der Bundeswehr ein zweistufiges SARS-CoV-2-Detektionsverfahren entwickelt, welches aus einem Screening- und Bestätigungsassay einschließlich simultanem Nachweis zweier interner Validitätskontrollen besteht. Im Rahmen des Validierungsprozesses konnte zudem eine robuste gefriergetrocknete RT-PCR-Chemie identifiziert werden, so dass dieser Inhouse-Assay auch als Back-up-Methode für den Auslandseinsatz zur Verfügung steht. Außerdem können dank des harmonisierten Temperaturprofils sowohl Screening- als auch Bestätigungsassay in einem PCR-Lauf durchgeführt werden, wodurch eine schnelle Diagnostik möglich ist.
Dieser Inhouse-Assay wurde nach nur drei Wochen intensiver Validierungsarbeit am ZInstSanBw Kiel in Kronshagen als Back-up-Methode dem BwKrhs Berlin zur Verfügung gestellt. Aufgrund der in der Zwischenzeit deutlich gebesserten Lieferbarkeiten kommerzieller Testkits und der Tatsache, dass in der Abteilung XXI BwKrhs Berlin als einem nach DIN 15189 akkreditierten Labor grundsätzlich CE-IVD-gekennzeichneten Testkits der Vorzug gegeben werden sollte, kam diese Methode schließlich nicht zum „scharfen“ Einsatz. Sie steht jedoch weiterhin für den Fall zur Verfügung, dass erneute internationale Lieferengpässe entstehen. Dieses Beispiel verdeutlicht sehr gut die hohe Dynamik der Lageentwicklung in dieser Zeit und unseren gemeinsamen, approbationsübergreifenden Ansatz, für alle Eventualitäten bestmöglich gerüstet zu sein.
Anpassung der Laborsoftware
Im Hinblick auf einen Einsatz in der Routinediagnostik fehlte noch ein wesentlicher Aspekt: die Anbindung an die vorhandene Labor-EDV zur fehlersicheren Übernahme der Messergebnisse bei den erwarteten hohen Probenzahlen. Hier zeigte sich erneut, wie schnell Lösungen gefunden werden, wenn alle Beteiligten konstruktiv und zielorientiert zusammenarbeiten. Während die Herstellerfirma der in den Abteilungen XXI verwendeten Laborsoftware, IMP Computersysteme AG, bereits mit einem mündlichen Auftrag die Einrichtung einer Schnittstelle zwischen Analysesystemen und Labor-IT vorbereitete, initiierte der S4-Bereich BwKrhs Berlin das offizielle Beschaffungsverfahren und beteiligte sich mit dem Teilbereich Medizintechnik zusammen mit einer externen Firma an der Einbindung in das Medizingerätenetz. Abteilung S6 BwKrhs Berlin stellte zusätzliche APC zur Verfügung. Die Konfiguration der Analyseparameter erfolgte durch den Erstautor dieses Beitrags als Systemadministrator der Laborsoftware IMP::Mikrobiologie.
Einsatzbereit nach nur 3 Wochen
Nach erfolgreicher Durchführung aller Tests und nach Probedurchläufen im Routineverfahren konnte ab dem 6. April 2020, also genau 3 Wochen nach Beginn der ersten Vorbereitungen, die SARS-CoV-2-PCR als Bestandteil der Routinediagnostik im BwKrhs Berlin angeboten werden. Mit dieser Methodik und dem genannten Personal wurde die Diagnostik bis Ende Juni fortgeführt und in der Kapazität auf 100-200 Untersuchungen pro Tag ausgeweitet. Aufgrund einer von uns initiierten Beschaffungsinitiative, welche durch Kdo SanDstBw nachdrücklich unterstützt und durch das Bundesamt für Ausrüstung, Informationstechnik und Nutzung der Bundeswehr sehr schnell umgesetzt wurde, stehen inzwischen in allen Abteilungen XXI Bw(Z)Krhs neue, vollautomatisierte Plattformen für die SARS-CoV-2-PCR-Diagnostik zur Verfügung.
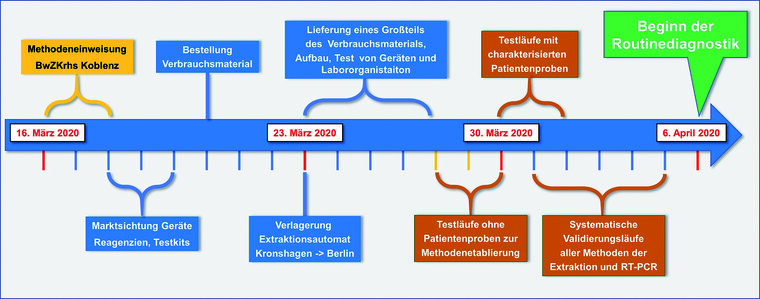
Abb. 1: Nur 3 Wochen wurden von der ersten Einweisung bis zum Beginn der Routinediagnostik „RT-PCR SARS-CoV-2“ benötigt.
Fazit
Die positivste Erfahrung im gesamten Prozess war die Zusammenarbeit im „Labor-Team Corona“. Das knapp bemessene Stammpersonal der Abteilung XXI sollte zunächst ausschließlich seine üblichen Aufgaben verrichten, die Besetzung des Coronavirus-PCR-Labors wurde nur durch externes Personal sichergestellt.
Begonnen hatte ein Viererteam mit dem Erstautor dieses Beitrags, Oberfeldveterinär Dr. Haarmann, die in der ASt Berlin des ZInstSanBw Kiel die Diagnostik leitet, der medizinisch-technischen Laboratoriumsassistentin (MTLA) Diana Grimm, die vom ZInstSanBw Kiel aus Kronshagen abgestellt wurde, und Oberstabsgefreiter Daniel Bartosik, einem Reservedienst leistenden Master in Molekularbiologie, der bereits häufiger in der Abteilung XXI Wehrübungen absolviert hatte. Wie an vielen anderen Dienststellen im Sanitätsdienst meldeten sich in der Krisensituation erfreulich zahlreiche weitere Reservisten zum Dienst und es wurde Personal aus der Truppe an die BwKrhs abgestellt. Im Verlauf kamen als Reservedienst Leistende ein promovierter Medizinwissenschaftler, zwei MTLA, ein promovierter Virologe und als Abstellung aus der Truppe zwei ärztliche Kollegen in Weiterbildung zum Laborarzt bzw. Mikrobiologen sowie ein Kollege aus der zwischenzeitlich betrieblich eingeschränkten Abteilung HNO des BwKrhs Berlin hinzu. So konnte eine durchhaltefähige Besetzung an 7 Tagen pro Woche gewährleistet werden. Wesentlich heterogener „zusammengewürfelt“ kann ein Team kaum sein – vielleicht war das auch ein Vorteil. Die Stimmung war gut und alle waren ausgesprochen motiviert, gemeinsam das Ziel zu erreichen.
Es heißt oft etwas despektierlich „typisch Bundeswehr“, wenn es darum geht, dass Flugzeuge nicht fliegen oder Panzer nicht fahren. Das hier Geschilderte ist meines Erachtens aber ebenfalls ein Beispiel für „typisch Bundeswehr“:
Wenn seitens der Führung den Mitarbeiterinnen und Mitarbeitern Vertrauen entgegengebracht wird, wenn damit formale Zwänge zugunsten kreativer und unkomplizierter Lösungen aufgegeben werden und Experten in einem unkonventionellen Rahmen Gelegenheit gegeben wird, ihr Fachwissen und ihre Erfahrungen „ungefiltert“ in ein Projekt einzubringen, dann können Herausforderungen bestanden werden, deren Bewältigung unter „normalen“ Rahmenbedingungen unmöglich erscheint. In diesem Sinne gebührt allen Beteiligten Dank, ein Projekt zu einem vorläufigen erfolgreichen Abschluss gebracht zu haben, dessen Umsetzung wenige Wochen zuvor noch als unrealistisch erschienen war.
Der Erfolg des Projekts „Implementierung virologischer Spezialdiagnostik im klinisch-mikrobiologischen Umfeld“ kann aber nur dann von Dauer sein, wenn es gelingt, die etablierte Fähigkeit durch geeignete Ertüchtigungsmaßnahmen zu konsolidieren. Denn auch dies gehört zu den „Lessons learned“:
Nach der Pandemie ist vor der Pandemie!
Für die Verfasser
Oberfeldarzt Dr. Werner Wenzel
Bundeswehrkrankenhaus Hamburg
Abteilung XXI – Mikrobiologie, Krankenhaushygiene
Lesserstraße 180, 22049 Hamburg
E-Mail: wernerwenzel@bundeswehr.org
1 Ein die Durchführung einer Real-time-PCR erklärendes (englischsprachiges) Video finden Sie unter https://www.youtube.com/watch?v=ThG_02miq-4.