PAUL-SCHÜRMANN-PREIS 2020
Bioforensische Untersuchungen an historischen Milzbrandproben als Grundlage für moderne Ausbruchsaufklärung im medizinischen B-Schutz
Bioforensic investigations on historical anthrax samples as a basis for modern outbreak reconnaissance in medical biodefense
Peter Brauna
a Institut für Mikrobiologie der Bundeswehr, München
Zusammenfassung
Hintergrund: Der Erreger des Milzbrands, Bacillus anthracis, und insbesondere seine Sporen zählen zu den berüchtigsten potenziellen Biokampfstoffen. Nur durch eine schnelle und exakte Aufklärung sowie Rückverfolgung kann bei Milzbrandausbrüchen zwischen einem natürlichen Auftreten des Erregers oder einer möglicherweise absichtlichen Ausbringung sicher unterschieden werden. Hierfür muss zuvor eine flächendeckende Phylogeografie des Erregers bekannt sein. In Ländern wie Deutschland, in denen Milzbrand nicht mehr oder nur sehr selten vorkommt, bestehen hier erhebliche Wissenslücken, die durch Rückgriff auf historische Proben früherer Milzbrandausbrüche zumindest teilweise geschlossen werden könnten.
Während Genomanalysen rezenter B. anthracis Isolate bereits eine eingehende phylogeografische Abdeckung in Regionen mit regelmäßigen natürlichen Milzbrandausbrüchen ermöglichen, fehlen Einblicke in die historische Genomik des Erregers für die Zeit vor Mitte des 20. Jahrhunderts, als Milzbrand auch in Mitteleuropa noch weit verbreitet war. Aufgrund dieser Datenlücke kann bisher nur sehr schwer entschieden werden, ob es sich bei den seltenen, neu auftretenden Milzbrandfällen in Deutschland um einen natürlichen Ausbruch oder eine beabsichtigte Ausbringung handelt.
Material und Methode: In dieser Arbeit wurde das bislang älteste dokumentierte B. anthracis Genom charakterisiert. Das inaktive 142 Jahre alte Material stammt von einem 1878 in Chemnitz verendeten Rind. Die DNA wurde mit einer speziell für die Analyse von historischen Präparaten entwickelten Methode extrahiert.
Ergebnisse: Das sequenzierte Genom mit 242 einzigartigen Basenpositionen gegenüber seinem nächsten rezenten Verwandten konnte in eine für Mitteleuropa als typisch postulierte, phylogenetische Hauptgruppe von B. anthracis („B. Branch CNEVA“) eingeordnet werden.
Diskussion und Schlussfolgerungen: Die Ergebnisse stützen die Hypothese, dass diese CNEVA-Gruppe einen Teil der einheimischen genetischen Abstammung von B. anthracis in Mitteleuropa bildet. Im Falle eines zukünftigen Ausbruchs wäre aus biologisch-forensischer- und wehrmedizinischer Sicht die Isolierung eines B. anthracis Stammes aus dieser Gruppe weit weniger alarmierend als ein Isolat aus einer Gruppe, für die es keine Belege für ein natürliches Vorkommen in Deutschland gibt. Damit ermöglicht der Blick in die Vergangenheit des potenziellen B-Kampfstoffs B. anthracis heute exaktere Ausbruchsaufklärung und bereitet so den medizinischen B-Schutz auf mögliche biologische Gefahrenlagen in der Zukunft vor.
Stichworte: Bacillus anthracis, Milzbrand, Anthrax, Sequenzierung, historisches Genom, Phylogenetik, Bioforensik, Medizinischer B-Schutz.
Summary
Background: The anthrax pathogen Bacillus anthracis and especially its spores are among the most notorious agents that can be misused for biological warfare or bioterrorism. For anthrax outbreaks, rapid and accurate reconnaissance as well as trace-back analyses are crucial to be able to differentiate between a natural occurrence of the pathogen and a possibly deliberate release. For this, a world-wide phylogeography of the pathogen must be available. However, in countries such as Germany, where anthrax outbreaks are very rare, little is known about the naturally occurring genotypes of the pathogen in the past.
While genomic analysis of recent B. anthracis isolates already provides in-depth phylogeographic coverage of regions with high frequencies of natural anthrax outbreaks, historical genomics of the pathogen for the period before the mid-20th century, a time when the pathogen was still wide-spread, are lacking. Due to this lack of data, it has been very difficult to determine whether rare, new emerging cases of anthrax in Germany are a natural outbreak or an act of intentional release.
Material and Methods: In this work, the oldest documented B. anthracis genome to date was characterized. The inactive 142 years old material had been taken from a cattle with anthrax disease in Chemnitz in 1878. DNA was extracted using a method specifically developed for the analysis of historical specimens.
Results: The sequenced genome features 242 unique base positions compared to its closest recent relative and belongs to a major phylogenetic group of B. anthracis (“B. Branch CNEVA”) postulated as typical for Central Europe.
Discussion and conclusions: The results support the hypothesis that this CNEVA group represents a native genetic lineage of B. anthracis in this part of Europe. In the event of a future outbreak, isolation of a B. anthracis strain from this group would raise, from a bioforensic perspective, far less concern than an isolate from a group for which there is no evidence of natural occurrence in Germany. Thus, looking into the past of the potential biological warfare agent B. anthracis allows for more accurate trace-back analyses of outbreaks today and thus prepares medical biodefense for potential biothreat situations in the future.
Keywords: Bacillus anthracis, anthrax, sequencing, historical genome, phylogenetics, bioforensics, medical biodefense.
Einleitung
Erreger
Bacillus anthracis, der Erreger des Milzbrands, ist ein Gram-positives, stäbchenförmiges, aerobes Bakterium, das bei widrigen Umweltbedingungen Sporen (Endosporen) bildet (Abbildung 1). Das Ausbilden von Sporen aus dem vegetativen Stadium der Bazillen wurde erstmals von Ferdinand J. Cohn beschrieben. Bereits zwei Jahre später (1876) untersuchte Robert Koch in seinem Werk „Die Ätiologie der Milzbrand-Krankheit“ den Lebens- und Infektionszyklus von B. anthracis. Darin wies er nach, dass das Bakterium als Dauerform Sporen ausbildet und sich daraus wieder vegetative Zellen bilden und es im geeigneten Wirt zur Erkrankung kommt. Damit widerlegte er Casimir Davaines These, wonach Milzbrand nur durch die als Stäbchen vorhandenen Bakterien verursacht wird [31].
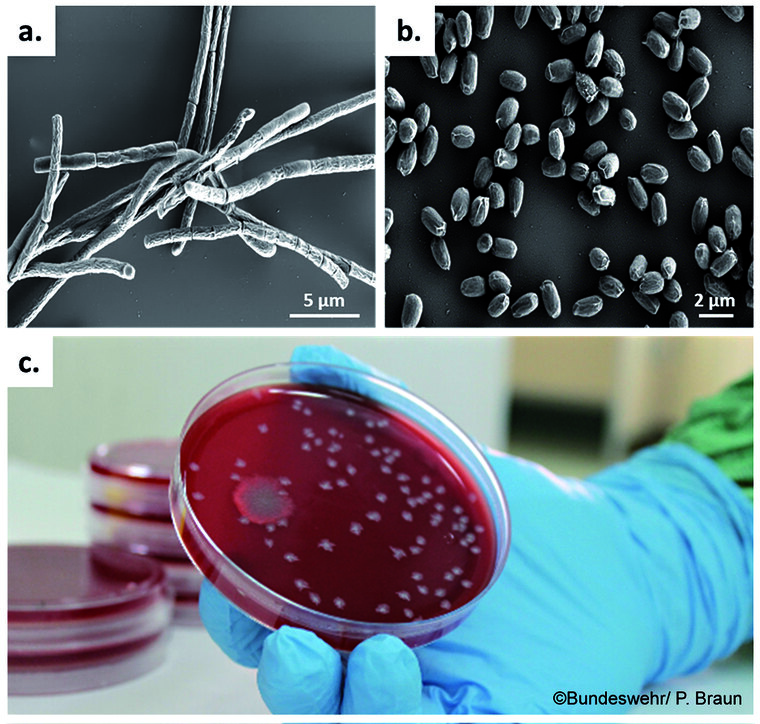
Abb. 1: B. anthracis ist ein stäbchenförmiger, Gram-positiver Sporenbildner. Elektronenmikroskopische Aufnahme von (a.) B. anthracis Zellen, die in Ketten wachsen und von (b.) B. anthracis Sporen. (c.) Weißlich-milchige, nicht hämolytische Kolonien von B. anthracis auf einer Blutagar- Platte zeigen die typische „Medusenkopf“-Form (neben einer flächig wachsenden, hämolytischen Kolonie des nahe verwandten Bacillus mycoides).
Infektionsmanifestationen
Milzbrand befällt Wild- und Weidetiere wie vorwiegend Rinder, Schafe, Ziegen oder Pferde. Menschen sind ebenfalls anfällig, benötigen aber höhere Infektionsdosen, damit sich die Krankheit manifestieren kann [36]. Die häufigste Form beim Menschen ist der Hautmilzbrand, der mehr als 95 % aller Anthrax-Fälle ausmacht. Hier geht man davon aus, dass die inerten Sporen nach der Exposition, z. B. mit infektiösem tierischem Gewebe, durch Mikroläsionen in die menschliche Haut eindringen und – nach der Aufnahme in Wirtsmakrophagen – in diesen auskeimen und so eine Infektion verursachen. Der Begriff Anthrax, griechisch für Kohle, leitet sich von den schwarzen Milzbrandkarbunkeln der Hautnekrose ab, die zusammen mit lokalen Ödemen die charakteristischen Erscheinungsformen des Hautmilzbrandes sind [36]. Historisch gesehen traten Infektionen dieser relativ milden Form (unbehandelt etwa 5–20 % Sterblichkeitsrate) typischerweise in Gerbereien oder wollverarbeitenden Betrieben auf, wo die Arbeiter mit kontaminierten Häuten in Kontakt kamen. Selten, aber mit einer aufgrund der hohen Wahrscheinlichkeit einer lymphatischen oder hämatogenen Ausbreitung höheren Sterblichkeitsrate von mehr als 50 % (selbst bei Behandlung), tritt Darmmilzbrand als Folge des Verzehrs von Fleisch infizierter Tiere auf [26][36]. Die gastrointestinale Form wird heute vorwiegend mit enzootischen Regionen in ländlichen und weniger entwickelten Gebieten in Verbindung gebracht, in denen Menschen in unmittelbarer Nähe zum Vieh leben und die tierärztliche Überwachung unzureichend ist [36]. Es wird geschätzt, dass in Afrika, Zentral- und Südasien bis zu zehn Fälle von Haut- und Darmmilzbrand beim Menschen nach der Schlachtung eines einzigen infizierten Tieres auftreten [36].
Milzbrand plagt den Menschen, Wild- und Nutztiere seit vielen hunderten, möglicherweise mehreren tausend Jahren [11]. Auch heute noch grassiert der Erreger in zahlreichen Regionen auf allen Kontinenten mit Ausnahme der Antarktis [36]. Nur wenigen Ländern ist es bis heute gelungen, durch staatliche und institutionelle Impfkampagnen, Überwachungsprogramme und Kampagnen zur sicheren Beseitigung von Kadavern wiederkehrende Milzbrandausbrüche auszumerzen. Selbst Deutschland, das in den letzten Jahren nur sehr begrenzte Ausbrüche (2009, 2012 und 2014) bei Nutztieren zu verzeichnen hatte [2][3][12], war noch vor einem Jahrhundert schwer von der Krankheit befallen. Von 1912–1932 (keine Daten für 1926) infizierten sich 2.518 Menschen, von denen 431 starben. Im gleichen Zeitraum erkrankten etwa 90.000 Nutztiere an der Krankheit [17]. Neben den natürlichen enzootischen und zoonotischen Manifestationen hat sich in Europa nach der Jahrtausendwende als neue Form der Injektionsmilzbrand manifestiert, bei dem sich Heroinkonsumenten höchstwahrscheinlich durch mit B. anthracis kontaminierte Heroinchargen infizierten [15]. Die vierte und schwerste Form von Milzbrand ist der Lungenmilzbrand, der nach Inhalation von B. anthracis Endosporen zu einer systemischen Infektion ähnlich dem Darmmilzbrand führt und unbehandelt in fast 100 % aller Fälle tödlich endet. Selbst mit Antibiotikatherapie liegt die Sterblichkeitsrate bei etwa 45 % [36].
Potenzieller biologischer Kampfstoff
Aufgrund seiner Virulenz und leichten Kultivierbarkeit und weil die Sporen von B. anthracis in der Umwelt sehr stabil und leicht herstellbar sind, gilt B. anthracis als der Erreger mit dem höchsten Potenzial, als biologischer Kampfstoff oder für bioterroristische Zwecke missbraucht zu werden. Er wird daher vom „Center for Disease Control and Prevention“ (CDC, USA) als Kategorie A-Erreger gelistet. Dieses Potenzial zeigte sich bei den Milzbrand-Anschlägen (Amerithrax) im Jahr 2001, als mit Sporen befüllte Briefe an mehrere Adressaten in den Vereinigten Staaten geschickt wurden, was zu 11 Fällen von Lungenmilzbrand und 11 Fällen von Hautmilzbrand führte. Fünf Personen starben an den Folgen der Infektion [14][18]. Es wird angenommen, dass die Freisetzung von B. anthracis Sporen in größeren Mengen (100 kg) über einem Ballungsgebiet bis zu 3 Millionen Menschen töten könnte. Dieses Szenario stellte das Potenzial eines Zusammenbruchs des zivilen Gesundheitssystems nach einem massiven Angriff mit Anthrax-Sporen eindrucksvoll dar [10]. Bereits 1971 wurde die „Konvention über das Verbot der Entwicklung, Herstellung und Lagerung bakteriologischer (biologischer) Waffen und Toxinwaffen sowie über die Vernichtung solcher Waffen“ von den Vereinten Nationen verabschiedet, nachdem es trotz des Genfer Protokolls zur Forschung und Anwendung von biologischen Waffen in der Zeit des zweiten Weltkrieges und des Kalten Krieges kam [7]. So ereignete sich z. B. 1917 der „Spionagezwischenfall mit Baron von Rosen“, bei dem Zuckerwürfel, die angeblich mit dem Milzbranderreger versetzt waren, verwendet werden sollten, um die alliierte pferdebetriebene Kriegsunterstützung zu sabotieren [4][28]. Zur Zeit des kalten Krieges (1979) kam es zu einem Vorfall in einer mutmaßlichen Biowaffenfabrik, bei dem große Mengen Milzbrandsporen entwichen und sich mit dem Wind über dem Stadtgebiet von Swerdlowsk (heute Jekaterinburg) verbreiteten. Insgesamt kam es dabei offiziell zu 66 Todesopfern in der Stadt [29]. Spätere genomische Analysen von Gewebeproben infizierter Personen ergaben, dass es sich bei dem Ausbruchsstamm nicht um einen gentechnisch veränderten Organismus handelte, sondern um einen Genotyp, wie er natürlicherweise auch in Russland vorkommt [29].
Aufgrund der Historie und des enormen Potenzials von B. anthracis, auch in Zukunft als biologischer Kampfstoff missbraucht zu werden, ist es für den medizinischen B-Schutz und damit auch für die Wehrmedizin notwendig, Fähigkeiten und Wissen vorzuhalten, die für eine schnelle und genaue Ausbruchsaufklärung nötig sind. So müssen u. a. detaillierte Informationen über die genetische Diversität und die Phylogenie des Erregers vorliegen [1].
Differenzierung durch Genotyp
B. anthracis ist ein sehr klonaler Organismus mit einer geringen Intraspezies-Diversität auf genomischer Ebene. Seine Evolution ist wahrscheinlich auf die begrenzten Reproduktionsphasen von 20–40 Generationen während der Wirtsinfektion beschränkt, während die daraus resultierenden Sporen jahrelang inaktiv in der Umwelt ruhen können [19]. Vergleicht man B. anthracis Isolate aus verschiedenen Teilen der Welt, wird seit dem ersten Auftreten als Krankheitserreger vor vermutlich etwa 3 000–6 000 Jahren [37] nur ein geringes Maß an genetischen Variationen beobachtet [20]. Die Analyse zahlreicher Genomdaten ergab jedoch, dass sich Stämme von B. anthracis in verschiedene phylogenetische Gruppen einteilen lassen. Für jede dieser Gruppen wurde eine spezifische Auswahl an alternativen Einzel-Nukleotidbasen-Positionen in verschiedenen Genomen bestimmt (sogenannte kanonische „Single-Nucleotide-Polymorphisms“ (SNPs)), anhand derer ein Stamm in eine bestimmte verwandtschaftliche Gruppe eingeordnet werden kann (Abbildung 2).
Ausgehend von den drei Hauptgruppen A, B und C lassen sich verschiedene B. anthracis-Isolate in 13 klassische „kanonische“ SNP- (canSNP) Gruppen unterteilen, welche die globalen phylogenetischen Beziehungen zwischen allen B. anthracis-Stämmen widerspiegeln [25][37] (Abbildung 2). Dieses Typisierungssystem wird heute oft für die epidemiologische Untersuchung von Ausbrüchen sowie für Rückverfolgungsanalysen in der Bioforensik verwendet und wurde z. B. beim Amerithrax-Fall 2001 eingesetzt, um den Ursprung des missbräuchlich verwendeten B. anthracis-Isolates zu ermitteln [16][19].
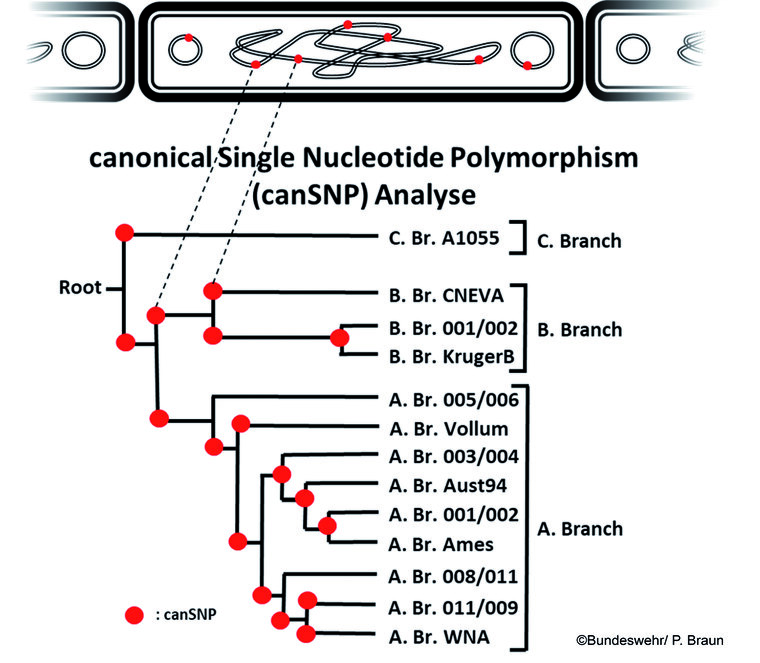
Abb. 2: Das bioforensische Instrument der „canonical Single Nucleotide Polymorphism“ (canSNP)-Analyse dient der Einordnung von B. anthracis-Stämmen in phylogenetische Verwandtschaftsgruppen und wird heute oft bei epidemiologischen Untersuchungen verwendet. Der obere Teil der Abbildung zeigt vereinfachte, in Ketten wachsende B. anthracis Zellen. Die Virulenzplasmide pXO1 und pXO2 (Kreise) und das Chromosom (verdrehter Kreis) sind repräsentativ mit canSNP-Positionen (rote Punkte) gekennzeichnet, die phylogenetisch relevant sind. Darunter sind ausgehend vom phylogenetischen Ursprung (letzter gemeinsamer Vorfahre: „Root“) die 13 Gruppen („Branches“) der drei Hauptlinien A, B und C abgebildet.
Phylogeografische Daten erforderlich
Um im Falle eines Milzbrandausbruchs schnell in der Lage zu sein, den Ursprung des Ausbruchs zu identifizieren und damit auch aufzuklären, ob es sich um einen natürlichen, also von Sporen aus der Umwelt hervorgerufenen, Ausbruch oder um eine absichtliche bzw. unabsichtliche Freisetzung des Erregers handelt, müssen genaue und flächendeckende phylogeografische Daten des Erregers zur Verfügung stehen. Um dies abschätzen zu können, muss also für jede Region der entsprechende natürlich vorkommende (autochtone) Genotyp bekannt sein. So würde beispielsweise ein Milzbrandausbruch im Krüger Nationalpark in Südafrika keine Indizien für eine absichtliche Freisetzung des Erregers liefern, wenn die Infektionen durch einen B. anthracis-Stamm der B. Branch Gruppe KrugerB [30] hervorgerufen würde. Stämme dieser Gruppe gehören hier der autochtonen B. anthracis-Population an. Hingegen würde ein Stamm aus der A. Branch Gruppe Ames, zu der u. a. der Stamm aus den Briefanschlägen aus dem Jahr 2001 gehört, starke Indizien für eine absichtliche Ausbringung liefern.
Um Informationen über die autochtonen, phyologenetischen Gruppen von B. anthracis in Ländern wie Deutschland zu erhalten, in denen Milzbrand nicht mehr oder nur noch sehr selten vorkommt, muss auf historische Proben und, soweit zugänglich, die wenigen vorhandenen Lebendisolate aus Stammsammlungen zurückgegriffen werden. Für andere berüchtigte Krankheitserreger wie Yersinia pestis (Pest) oder Mycobacterium tuberculosis (Tuberkulose) gibt es umfangreiche Informationen über die historische Phylogeographie der Erreger aus menschlichen (oder tierischen) Überresten. So ist beispielsweise der älteste molekulare Nachweis für die historische Pest aus Schweden 4 900 Jahre alt [27]. Dagegen reichen unsere Kenntnisse über die historische Phylogenie von B. anthracis kaum mehr als hundert Jahre zurück. Obwohl Louis Pasteur sein berühmtes öffentliches Anthrax-Impfexperiment 1881 durchführte, begann die Collection de l‘Institut Pasteur (CIP) erst 1892 mit der Sammlung von Bakterienstämmen [8]. Diese Stämme wurden jedoch seither wiederholt kultiviert und haben sehr wahrscheinlich über die Zeit genetische Veränderungen angesammelt. Umgekehrt sind tote Exemplare oder Bakterien aus menschlichen Überresten evolutionär inert.
Während es in der Kultur eine Anzahl lebender mitteleuropäischer B. anthracis-Isolate aus der relativ jungen Geschichte (ab ca. 1950) gibt, stammt das älteste in der Literatur erwähnte Isolat aus der Zeit um 1890 [39]; es fehlen jedoch die zugehörigen Metadaten. Daher bleibt sein Ursprung schwer erfassbar. Kürzlich wurde B. anthracis aus Ausgrabungsstätten in der Permafrostzone im Norden Russlands isoliert [34]. Ob diese Isolate jedoch tatsächlich von tausende Jahre alten Kadavern stammten, bleibt fraglich. Es kann nicht ausgeschlossen werden, dass diese Sporen aus jüngeren Ausbrüchen tiefere, ältere Bodenhorizonte kontaminiert haben. Nichtsdestotrotz könnten angesichts des Auftauens großer Permafrostgebiete in hohen Breitengraden vermehrt Milzbrandausbrüche bevorstehen [34].
Analyse historischer Proben
Obwohl historische Exemplare von B. anthracis (z. B. fixiert auf Glasobjektträgern) ein wertvolles Mittel zur Erweiterung unseres Wissens über die Vielfalt der Anthrax-Erreger sind, gibt es bisher nahezu keine Studien hierzu – obgleich solche Präparate existieren. Veröffentlichungen enthalten nur Informationen aus relativ „jungen“ Proben: ein paar Jahre alte Proben aus Jordanien [5], bis zu 30 Jahre alte Proben aus Sambia [13] oder etwa 35 Jahre alte, in Paraffin eingebettete Proben aus der ehemaligen Sowjetunion [29].
Somit gibt es bis heute kein beschriebenes, wirklich historisches Genom von B. anthracis. In dieser Arbeit wurde, ausgehend von einem Objektträger von 1878, das älteste dokumentierte und somit erste echte historische Genom von B. anthracis bioinformatisch rekonstruiert und charakterisiert.
Methoden
B. anthracis-Stämme, Wachstumsbedingungen und Extraktion von DNA aus inaktiviertem Kulturmaterial
Alle in dieser Studie verwendeten Stämme (Genome) sind in der Tabelle 1 aufgeführt. B. anthracis-Kulturen aus der Stammsammlung des Instituts wurden im Labor der Biosicherheitsstufe 3 auf Blutagar kultiviert und dann vor der weiteren Verwendung chemisch inaktiviert [9]. Die DNA aus inaktivierten Zellen wurde mit dem MasterPure™-Gram-Positive-DNA-Purification Kit (Lucigen) isoliert und die DNA-Konzentrationen mit dem Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific) nach den Protokollen der Hersteller quantifiziert. DNA-Proben wurden bis zur weiteren Verwendung bei -20°C gelagert.
Tab.1: Datenbankeinträge und Metadaten der Genomsequenzen neu sequenzierter und zusätzlicher B. anthracis-Stämme aus öffentlich zugänglichen Repositorien
*Isolationsjahr unbekannt
Mikroskopische Auswertung der historischen B. anthracis-Probe
Der Original-Objektträger mit einem B. anthracis Blutausstrich wurde sorgfältig aus den Verpackungsumschlägen ausgepackt und mittels Phasenkontrastmikroskopie (630-fache Vergrößerung) unter Verwendung eines Leica DMi8-Mikroskops untersucht.
DNA Extraktion vom historischen Objektträger
Um eine Kontamination zu vermeiden wurden alle Manipulationen im Zusammenhang mit der Probe Chemnitz 1878 (M-0290509, veröffentlicht 1879 als Nr. 700 der Exsiccatae Serie [33]) in einem Labor durchgeführt, das zuvor nicht für den Umgang mit B. anthracis-DNA genutzt wurde.
Mit einem sterilen Tupfer (Copan Nylon Floq Swab, Hain Life Science), der mit 100 µL steriler phosphatgepufferter Kochsalzlösung befeuchtet wurde, wurde vorsichtig ein Teil des Blutausstrichs entnommen, indem der Tupfer langsam gedreht und die Oberfläche in einer zickzack-ähnlichen Bewegung beprobt wurde. Der Tupfer wurde 15 min lang unter einem laminaren Luftstrom luftgetrocknet, der Tupferkopf abgeschnitten und in ein 2 mL-Mikrozentrifugenröhrchen gegeben. Die DNA wurde unter Verwendung des MasterPure™ Gram-positive DNA-Reinigungskits (Lucigen) nach einem modifizierten Protokoll extrahiert. Hierzu wurden 150 µL TE-Puffer mit 1250 U Ready-Lyse-Lysozymlösung direkt auf den Tupferkopf gegeben und bei 37°C für 60 min inkubiert. Dazu wurde 1 μL Proteinase K (50 μg/μL), verdünnt in 150 μL grampositive lysis solution, zugegeben. Das Röhrchen wurde dann bei 65°C für 15 min bei 900 U/min inkubiert und alle 5 min kurz gemischt. Nachdem die Probe 5 min lang auf Eis gelegt worden war, wurden die Flüssigkeit und der Tupferkopf in eine QIAshredder Zentrifugationssäule überführt und 2 min lang bei 14 000 U/min zentrifugiert. Der Tupferkopf wurde verworfen, dem Durchfluss wurden 175 µL des ‹MPC› Protein Precipitation Reagent hinzugefügt. Nach mehrfachem Durchmischen wurden die Zelltrümmer durch Zentrifugation bei 4°C für 10 min bei 14 000 U/min in einer Mikrozentrifuge pelletiert. Der Überstand wurde in ein frisches Mikrozentrifugenröhrchen überführt. Dazu wurden 5 µL Roti-Pink (Roth), 10 µL Glykogenlösung (5 mg/mL, Roth) und 500 µL Isopropanol (Roth) gegeben und durch 30–40-maliges Invertieren des Röhrchens vorsichtig gemischt. Die DNA wurde durch Zentrifugation bei 4°C für 10 min 14 000 U/min in einer Mikrozentrifuge pelletiert. Das DNA-Pellet wurde zweimal mit 200 µl 70 %-igem Ethanol gewaschen. Nach vollständigem Entfernen des Ethanols wurde die DNA in 50 µL TE-Puffer resuspendiert und bis zur weiteren Verwendung bei -20°C gelagert.
Sequenzierung des historischen Genoms und Datenanalyse
Aus der Gesamt-DNA wurde eine Illumina-kompatible Bibliothek hergestellt (NEBNext® Ultra™ II DNA Library Prep Kit, NEB) und auf einem MiSeq-Gerät (Illumina) unter Verwendung der MiSeq V3-Chemie sequenziert. Hochwertige „paired-end“ Reads wurden de novo unter Verwendung eines institutseigenen Skripts auf der Grundlage von SPAdes Assembler [6] und Pilon [40] assembliert. Um altersbedingte DNA-Sequenz-Artefakte auszuschließen und falsche Schlussfolgerungen zu vermeiden, wurde die Genomsequenz manuell wie folgt zusammengefügt:
- Zuerst wurden die erhaltenen „scaffolds“ (Software BWA-SW (Li and Durbin 2010)) mit dem Stamm BF-1, einem engen genetischen Nachbarn, verglichen („mapping“). Regionen, die durch keine Reads abgedeckt waren, wurden von der Konsensus-Sequenz ausgeschlossen.
- Zweitens wurde BWA-mem verwendet, um die Reads auf die geordneten Contigs umzuordnen.
- Drittens wurde Mpileup (Software-Paket SAMtools [23]) mit Standardparametern verwendet, um mehrdeutige Basenpositionen zu eliminieren.
Alle uneindeutigen Positionen (n = 9326, entsprechend 0,17 % des Chromosoms) wurden in der entsprechenden Konsensus-Fasta-Datei mit „N“ maskiert. Diese abschließend zusammengeführte Sequenz wurde für weitere vergleichende Analysen verwendet.
Für den mehrfachen chromosomenweiten SNP-Vergleich von B. anthracis wurde das Parsnp-Tool (Harvest Suite) verwendet [35]. Dazu wurden neben Chemnitz 1878 repräsentative B. anthracis-Genome aus öffentlichen Datenbanken (Tabelle 1) und neu sequenzierte Genome aus der Instituts-Sammlung (Tirol 3520 und 6282) unter Verwendung des B. anthracis Ames Ancestor Referenzchromosoms (NC_007530) als phylogenetische Außengruppe miteinander verglichen (Parsnp-Parameter -c -e -u -C 1000). SNPs wurden mit HarvestTools (Version 1.0) aus derselben Software-Sammlung [35] in eine „Multi Isolate“ vcf-Datei extrahiert. Um die Datenqualität zu verbessern, wurden eng benachbarte SNPs mit einem Abstand von weniger als 10 bp sowie Positionen, die undefinierte Nukleotide („N“) beherbergen, entfernt. Diese vcf-Datei wurde als Eingabedatei in HarvestTools verwendet, um eine FASTA-Datei mit den verketteten SNPs des untersuchten Chromosomensatzes als „Multiple Sequence Alignment“ zu erstellen. Diese verketteten Sequenzinformationen wurden zur Ableitung und Analyse („Verwandtschaftsbaum“) einer Maximum-Likelihood-Phylogenie in der Software MEGA 7 [21][32] verwendet. Ein „Minimum Spanning Tree“ wurde in BioNumerics 6.6 (Applied Maths) aus der vcf-SNP-Datei (im Binärformat) als Input berechnet und sein Stil manuell bearbeitet.
Ergebnisse und Diskussion
Analysiertes Präparat
In der Botanischen Staatssammlung München wurde bei einer Bestandsaufnahme im Jahr 2018 auf einem 142 Jahre alten Objektträger eine infizierte Blutprobe wiederentdeckt [33]. Der Objektträgerdeckel war detailliert beschriftet und gab Auskunft über den verantwortlichen Veterinär sowie den Zeitpunkt und den geografischen Standort des erkrankten Rindes, von dem die Blutprobe entnommen wurde (Abbildung 3a). Das Präparat wurde 1878 von Dr. O. E. R. Zimmermann angefertigt (Abbildung 3b), nur zwei Jahre nachdem Robert Koch mit der systematischen Erforschung von B. anthracis begonnen hatte und damit die moderne Mikrobiologie mitbegründete. Eine sorgfältige mikroskopische Dokumentation des Präparats durch den Autor zeigte stäbchenförmige Strukturen neben wahrscheinlich ausgetrockneten Rinderblutzellen und möglicherweise sogar Sporen, die die Vermutung stützten, dass B. anthracis tatsächlich vorhanden sein könnte (Abbildung 3c).
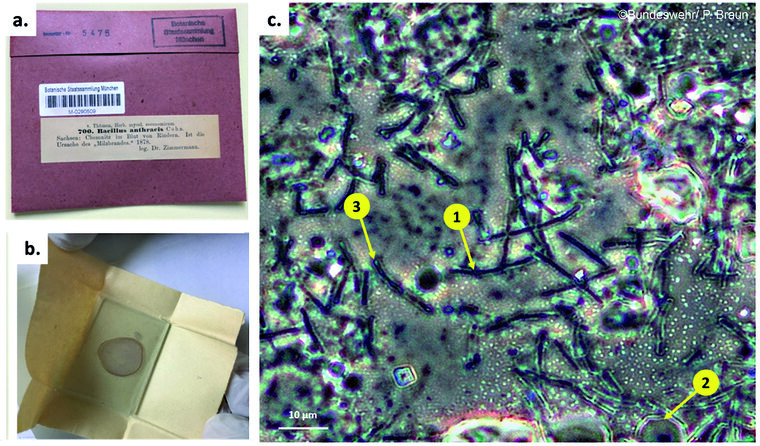
Abb. 3: Erste Charakterisierung eines B. anthracis-Exemplars von 1878:
(a.) Das Exemplar mit Original-Etikett („Sachsen: Chemnitz im Blut von Rindern. Ist die Ursache des „Milzbrandes“ 1878“)
(b.) Ausgepacktes, papierumwickeltes Präparat ohne Deckglas; die rote Farbe der Rinderblutzellen ist noch erkennbar.
(c.) Mikroskopische (Phasenkontrast-) Untersuchung der Probe: Pfeil #1 zeigt stäbchenförmige Strukturen, wahrscheinlich B. anthracis-Zellen. Pfeil #2 zeigt eine mögliche Rinderblutzelle und Pfeil #3 eine Struktur, die einer naszierenden Endospore ähnelt.
DNA-Sequenzierung
Mit Hilfe einer speziell für diesen Bedarf entwickelten Extraktionsmethode konnte DNA aus dem historischen Präparat isoliert werden. Überraschenderweise waren Qualität und Quantität der extrahierten DNA für die molekulargenetische Vorabanalyse (PCR) und die anschließende DNA-Sequenzierung des gesamten Genoms ausreichend. So wurden 2 x 21 106 786 Reads generiert. Von den sechs erhaltenen Gigabasen waren nur etwa 2 % (117 288 692 Basen) von B. anthracis. Die verbleibenden 98 % waren bakteriellen (z. B. Cutibacterium sp.) oder bovinen Ursprungs und 31 % (6 590 587 Reads) waren menschliche Kontaminationen. Letztere spiegeln wahrscheinlich den wiederholten Kontakt mit Museumspersonal während der letzten mehr als 100 Jahre der Lagerung wider, da der Objektträger nicht mit einem Deckglas geschützt war.
Die genomische in silico-Analyse ergab, dass der chromosomale B. anthracis-Marker dhp61 sowie Marker für beide Virulenzplasmide vorhanden waren. Durch die canSNP-Typisierung wurde das historische Genom, das „Chemnitz 1878“ genannt wurde, phylogenetisch in die B.Br.CNEVA-Gruppe von B. anthracis eingeordnet[37].
Genaue phylogenetische Einordnung
Als nächstes wurde die phylogenetische Einordnung des Genoms von Chemnitz 1878 innerhalb der B.Br.CNEVA Gruppe genauer untersucht (Abbildung 4a).
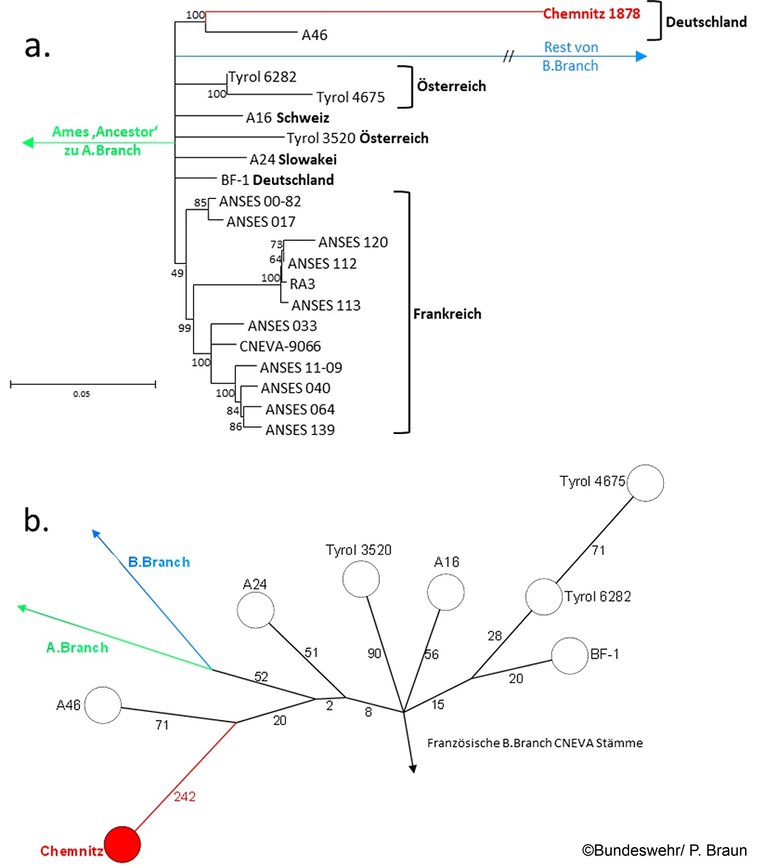
Abb. 4: Phylogenetische Einordnung von B. anthracis Chemnitz 1878. (a.) „Maximum Likelihood“ Baum, der von chromosomalen SNPs von Chemnitz 1878 und repräsentativen Verwandten abgeleitet wurde (insgesamt 2450 chromosomale SNPs; „Bootstrap“ statistische Vertrauenswerte basieren auf 500 Permutationen). Einzelne Namen und Herkunftsländer sind an den Zweigenden angegeben. Der Baum hat seine Wurzeln im B. anthracis Referenzstamm Ames ‚Ancestor‘, der zur A.Br.Ames Gruppe gehört.
(b.) „Minimum Spanning Tree“ auf der Grundlage chromosomaler SNPs, der den Stamm Chemnitz 1878 neben seinen nächsten Verwandten zeigt; Zahlen neben den Zweiglinien zeigen die Anzahl der SNPs an, die Knoten oder Stämme trennen.
Der nächste lebende Verwandte des Chemnitz 1878 Genoms war der Stamm A46, isoliert aus einem Schwein in der Nähe von Stuttgart mit einem Abstand von 313 SNPs, obwohl ein entfernterer Verwandter, das Rinderisolat BF-1 aus Bayern, einen Abstand von 307 SNPs hatte (Abbildung 4a). Zur gleichen Abstammungslinie gruppierten sich Isolate aus Mitteleuropa (Österreich, Schweiz und Slowakei). Von diesen wies der Stamm Tyrol 4675 die meisten SNP Unterschiede zum Stamm Chemnitz 1878 auf (386 SNPs; Abbildung 4b). B.Br.CNEVA-Stämme aus Frankreich gruppierten sich phylogenetisch weiter entfernt und bildeten mehrere unterschiedliche Sub-Cluster innerhalb der CNEVA canSNP-Gruppe (Abbildung 4a).
Während deutsche B. anthracis-Stammsammlungen eine breite Vielfalt von 8 der 12 ursprünglichen B. anthracis-canSNP-Gruppen aufweisen [37], deuten neuere Daten darauf hin, dass Isolate aus der B-Gruppe die autochthone Population des Anthrax-Erregers in Ländern aus Mittel- [38][39] und Nord- [24] Europa sowie aus Nordwestasien (Russische Föderation) bilden [34]. Diese Annahme wird durch die Ergebnisse dieser Arbeit stark unterstützt. Damit könnte aus bioforensischer und somit auch aus wehrmedizinischer Sicht bei einem zukünftigen Ausbruch nach Isolierung eines B. anthracis-Stammes aus der B.Br.CNEVA-Gruppe zunächst davon ausgegangen werden, dass es sich wahrscheinlich um eine natürliche, zufällige Infektion mit einem heimischen Milzbrand-Erreger handelt. Im Gegensatz dazu sollte die Isolierung von B. anthracis, der einer canSNP-Gruppe außereuropäischen Ursprungs zugeordnet werden kann, höchst alarmierend sein, da dies auf Bioterrorismus oder -kriminalität hindeuten würde. Somit ermöglicht der Blick zurück in die Vergangenheit des potenziellen B-Kampfstoffs B. anthracis eine genauere Bioforensik und bereitet den medizinischen B-Schutz auf mögliche Gefahrenlagen in der Zukunft vor.
Zusätzlich betont diese Arbeit, dass Sammlungsobjekte wie der hier untersuchte Glasobjektträger in historischen Sammlungen unschätzbar wertvolle Quellen für die Rekonstruktion der historischen Phylogenie des Milzbranderregers in Ländern sind, in denen die Krankheit heute praktisch ausgerottet ist. Eine breitere Bekanntmachung dieses Ansatzes würde wahrscheinlich auch solche Pannen (aus wissenschaftlicher Sicht) vermeiden, wie diejenige im Chrysler Herbarium an der Rutgers-Universität im Jahr 2016 (https://news.illinois.edu/view/6367/350255). Während eines Digitalisierungsprojekts historischer Proben wurde ein 121 Jahre altes Exemplar, ein Umschlag mit der Aufschrift B. anthracis, katalogisiert. Leider war der Inhalt des Umschlags bereits Jahre zuvor ohne weitere Charakterisierung vernichtet worden.
Literatur
- Antwerpen M, Beyer W, Bassy O, Ortega-García MV, Cabria-Ramos JC, Grass G, Wölfel R: Phylogenetic placement of isolates within the Trans-Eurasian clade A.Br.008/009 of Bacillus anthracis. Microorganisms 2019; 7(12): 689. mehr lesen
- Antwerpen M, Elschner M, Gaede W, Schliephake A, Grass G, Tomaso H: Genome sequence of Bacillus anthracis strain Stendal, isolated from an anthrax outbreak in cattle in Germany. Genome Announc 2016; 4(2): e00219–16. mehr lesen
- Antwerpen M, Proenca DN, Ruckert C et al.: Draft genome sequence of Bacillus anthracis BF-1, isolated from Bavarian cattle. J Bacteriol 2012; 194(22): 6360-6361. mehr lesen
- Antwerpen M, Sahl JW, Birdsell D et al.: Unexpected relations of historical anthrax strain. MBio 2017; 8(2): e00440-17. mehr lesen
- Aqel AA, Hailat E, Serrecchia L, Aqel S, Campese W, VicariN, Fasanella A: Molecular characterization of the circulating Bacillus anthracis in Jordan. Trop Anim Health Prod 2015; 47(8): 1621-1624. mehr lesen
- Bankevich A, Nurk S, Antipov D et al.: SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J Comput Biol 2012; 19(5): 455-477. mehr lesen
- Barras V, Greub G: History of biological warfare and bioterrorism. Clin Microbiol Infect 2014; 20(6): 497-502. mehr lesen
- Bennett R J, Baker KS: Looking backward to move forward: the utility of sequencing historical bacterial genomes. J Clin Microbiol 2019; 57(8): e00100-19. mehr lesen
- Braun P, Grass G, Aceti A et al.: Microevolution of anthrax from a young ancestor (M.A.Y.A.) suggests a soil-borne life cycle of Bacillus anthracis. PLoS ONE 2015;10(8): e0135346. mehr lesen
- Congress US: Assessment, Proliferation of Weapons of Mass Destruction, Proliferation of Weapons of Mass Destruction: Assessing the Risks. O. o. Technology. Washington, DC, U.S. Government Printing Office 1993.
- Dirckx JH: Virgil on anthrax. Am J Dermatopathol 1981; 3(2): 191-195. mehr lesen
- Elschner M C, Busch A, Schliephake A, Gaede W, Zuchantke E, Tomaso H: High-quality genome sequence of Bacillus anthracis strain 14RA5914 isolated during an outbreak in Germany in 2014. Genome Announc 2017; 5(40): e01002-17. mehr lesen
- Fasanella AL, Serrecchia L, Chiaverini A, Garofolo G, Muuka GM, Mwambazi L: Use of canonical single nucleotide polymorphism (canSNPs) to characterize Bacillus anthracis outbreak strains in Zambia between 1990 and 2014. PeerJ 2018; 6: e5270. mehr lesen
- Fowler RA, Shafazand S: Anthrax bioterrorism: prevention, diagnosis and management strategies. J Bioterr Biodef 2011; 2(107): doi:10.4172/2157-2526.1000107. mehr lesen
- Hanczaruk M, Reischl U, Holzmann T et al.: Injectional anthrax in heroin users, Europe, 2000-2012. Emerg Infect Dis 2014; 20(2): 322-323. mehr lesen
- Hoffmaster AR, Fitzgerald CC, Ribot E, Mayer LW, Popovic T: Molecular subtyping of Bacillus anthracis and the 2001 bioterrorism-associated anthrax outbreak, United States. Emerg Infect Dis 2002; 8(10): 1111-1116. mehr lesen
- Hunziker S: Über gewerblichen Milzbrand. Dissertation VetMed, Basel, 1939; Amriswil.
- Inglesby TV, O'Toole T, Henderson DA et al.: Anthrax as a biological weapon, 2002: updated recommendations for management. JAMA 2002; 287(17): 2236-2252. mehr lesen
- Keim P, Van Ert MN, Pearson T, Vogler AJ, Huynh LY, Wagner DM: Anthrax molecular epidemiology and forensics: using the appropriate marker for different evolutionary scales. Infect Genet Evol 2004; 4(3): 205-213. mehr lesen
- Keim PS, Wagner DM: Humans and evolutionary and ecological forces shaped the phylogeography of recently emerged diseases. Nature Reviews Microbiology 2009; 7(11): 813-821. mehr lesen
- Kumar S, Stecher G, Tamura K: MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol Biol Evol 2016; 33(7): 1870-1874. mehr lesen
- Li H, Durbin R: Fast and accurate long-read alignment with Burrows-Wheeler transform. Bioinformatics 2010; 26(5): 589-595. mehr lesen
- Li H, Handsaker B, Wysoker A et al.: The sequence alignment/map format and SAMtools. Bioinformatics 2009; 25(16): 2078-2079. mehr lesen
- Lienemann T, Beyer W, Pelkola K, Rossow H, Rehn A, Antwerpen M, Grass G: Genotyping and phylogenetic placement of Bacillus anthracis isolates from Finland, a country with rare anthrax cases. BMC Microbiol 2018; 18(1): 102. mehr lesen
- Marston CK, Allen CA, Beaudry J et al.: Molecular epidemiology of anthrax cases associated with recreational use of animal hides and yarn in the United States. PLoS One 2011; 6(12): e28274. mehr lesen
- Missiakas DM, Schneewind O: Bacillus anthracis and the pathogenesis of anthrax. In: Infectious Diseases: Biological Weapons Defense: Infectious Diseases and Counterterrorism (ed. by Lindler LE, Lebeda FJ, Korch GW). Totowa NJ, Humana Press Inc 2005: 79-97. mehr lesen
- Rascovan N, Sjogren KG, Kristiansen K et al.: Emergence and spread of basal lineages of Yersinia pestis during the Neolithic decline. Cell 2019; 176(1-2): 295-305 e210. mehr lesen
- Redmond C, Pearce MJ, Manchee RJ, Berdal BP: Deadly relic of the Great War. Nature 1998; 393(6687): 747-748. mehr lesen
- Sahl JW, Pearson T, Okinaka R et al.: A Bacillus anthracis genome sequence from the Sverdlovsk 1979 autopsy specimens. MBio 2016; 7(5): e01501-16. mehr lesen
- Smith KL, DeVos V, Bryden H, Price LB, Hugh-Jones ME, Keim P: Bacillus anthracis diversity in Kruger National Park. J Clin Microbiol 2000;38(10): 3780-3784. mehr lesen
- Sternbach G: The history of anthrax. J Emerg Med 2003; 24(4): 463-467. mehr lesen
- Tamura K, Nei M: Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol 2003; 10(3): 512-526. mehr lesen
- Thümen F: Herbarium mycologicum oeconomicum. Vienna (Austria), IndExs Exsiccatae 1879: ID 307188034.
- Timofeev V, Bahtejeva I, Mironova R et al.: Insights from Bacillus anthracis strains isolated from permafrost in the tundra zone of Russia. PLoS ONE 2019; 14(5): e0209140. mehr lesen
- Treangen TJ, Ondov BD, Koren S, Phillippy AM: The Harvest suite for rapid core-genome alignment and visualization of thousands of intraspecific microbial genomes. Genome Biol 2014; 15(11): 524. mehr lesen
- Turnbull PC: Anthrax in humans and animals - fourth edition. Geneva (CH), WHO Press 2008. mehr lesen
- Van Ert MN, Easterday RD, Huynh LY et al.: Global genetic population structure of Bacillus anthracis. PLoS One 2007; 2(5): e461. mehr lesen
- Vergnaud G: Bacillus anthracis evolutionary history: taking advantage of the topology of the phylogenetic tree and of human history to propose dating points." Erciyes Med J 2020; 42(4): 362-369. mehr lesen
- Vergnaud G, Girault G, Thierry S, Pourcel C, Madani N, Blouin Y: Comparison of French and worldwide Bacillus anthracis strains favors a recent, post-Columbian origin of the predominant North-American clade. PLoS One2016; 11(2): e0146216. mehr lesen
- Walker BJ, Abeel T, Shea T et al.: Pilon: an integrated tool for comprehensive microbial variant detection and genome assembly improvement. PLoS ONE 2014; 9(11): e112963. mehr lesen
Manuskriptdaten
Eingereicht: 29. Juni 2020
Angenommen: 21. September 2020
Zitierweise
Braun P: Bioforensische Untersuchungen an historischen Milzbrandproben als Grundlage für moderne Ausbruchsaufklärung im medizinischen B-Schutz. WMM 2021; 65(7): 264-271
Verfasser
Major Peter Braun
Institut für Mikrobiologie der Bundeswehr
Neuherbergstr. 111, 81677 München
E-Mail: peter3braun@bundeswehr.org
Die Arbeit wurde mit dem Paul-Schürmann-Preis 2020 der Deutschen Gesellschaft für Wehrmedizin und Wehrpharmazie e. V. ausgezeichnet.
Manuscript data
Submitted: June 29, 2020
Accepted: September 21, 2020
Citation
Braun P: Bioforensic investigations on historical anthrax samples as a basis for modern outbreak reconnaissance in medical biodefense. WMM 2021; 65(7): 264-271.
Author
Major Peter Braun
Bundeswehr Institute for Microbilogy
Neuherbergstr. 111, D-81677 Muenchen
E-Mail: peter3braun@bundeswehr.org
This study was honored by the German Society of Military Medicine and Pharmacy with the 2020 Paul-Schuermann-Award.