Pulse-Controlled Amplification – eine neuartige Methode für die Erregeridentifizierung mittels Nukleinsäurenachweis
(Vortrags-Abstract)
Katharina Müllera, Katrin Zwirglmaiera, Sarah Daßenb, Joachim Stehrb, Lars Ullerichb, Kilian Stoeckera
a Institut für Mikrobiologie der Bundeswehr, München
b GNA Biosolutions GmbH, Martinsried
Einleitung
Der Nachweis gefährlicher Infektionserreger ist für den medizinischen B-Schutz von herausragender Bedeutung. Diagnostischer Goldstandard ist dabei fast immer der Nukleinsäurenachweis mittels Polymerase-Kettenreaktion (PCR). Die PCR zeichnet sich dabei durch ihre hohe Sensitivität und Spezifität aus, gleichzeitig ist sie jedoch zeitaufwändig und durch hohen Materialaufwand laborgebunden. Eine schnelle und ortsunabhängige Alternative sollen Schnelltests bieten. Sie sind einfach durchzuführen und liefern Ergebnisse innerhalb von nur 15 min, es mangelt ihnen jedoch erheblich sowohl an Sensitivität als auch an Spezifität [6].
Als Schnittstelle zwischen PCR und Schnelltests haben wir deshalb eine neuartige und hochmobile Alternativmethode zur Erregeridentifizierung mittels Nukleinsäurenachweis, die sogenannte Pulse-Controlled Amplification (PCA), identifiziert und im Rahmen einer zivil-militärischen Zusammenarbeit gemeinsam mit einem Münchner Start-Up weiterentwickelt und getestet.
Die PCA beruht wie die PCR ebenfalls auf der Vervielfältigung von Nukleinsäuren, sie ist allerdings mit deutlich weniger Zeitaufwand verbunden. Die Schnelligkeit wird dadurch erreicht, dass das Reaktionsgemisch nicht in seiner Gesamtheit erhitzt und wieder abgekühlt wird. Stattdessen werden durch Golddrähte, die direkt in das Reaktionsgemisch eingebettet sind, kurze Stromimpulse geleitet. Durch diese Impulse werden sowohl der Draht als auch das Reaktionsvolumen erhitzt. Zwischen den einzelnen Stromimpulsen hingegen kühlen die Golddrähte und das Reaktionsgemisch nahezu augenblicklich wieder ab, was zu ultraschnellen Heiz- und Kühlzyklen führt und einen Nukleinsäurenachweis innerhalb kürzester Zeit ermöglicht. Durch die Verwendung von Sonden oder interkalierender Farbstoffe kann die Amplifikation außerdem in Echtzeit verfolgt werden. Gleichzeitig ist der Prototyp-Cycler (Pharos Micro), auf dem die PCA durchgeführt wird, klein, leicht und kann mittels Akku betrieben werden, was eine direkte Anwendung im Feld im Rahmen des medizinischen B-Schutzes ermöglicht (Abbildung 1).

Abb. 1: Prototyp-Cycler „Pharos Micro“
Methoden
Um die Leistungsfähigkeit dieser neuen Methode zu testen, wurden im Rahmen dieses Projektes zwei PCA-Assays unabhängig voneinander entwickelt und evaluiert. Der erste Assay wurde zum Nachweis von Yersinia pestis aus Umwelt- und Patientenproben unter spezieller Berücksichtigung der Feldtauglichkeit entworfen. Hierzu wurde, in Anlehnung an bereits etablierte PCR-Protokolle, das sogenannte pla-Gen als Zielsequenz identifiziert und entsprechende Primer und Sonden konzipiert [4][5]. Um das System von vornherein für die direkte Nutzung im Feld zu optimieren, wurde zudem ein gefriergetrockneter Mastermix sowie ein spezielles Zweistufenprotokoll, das einen Nachweis ohne vorherige Probenbearbeitung bzw. Nukleinsäureextraktion ermöglicht, entwickelt.
Der zweite Assay wurde im Rahmen der Corona-Pandemie zum schnellen Nachweis von SARS-CoV-2 aus Abstrichtupfern entwickelt. Dazu wurden die in der klassischen PCR eingesetzten Primer- und Sondensequenzen [1] für die PCA angepasst und optimiert und zum Nachweis der viralen RNA ein PCA-Assay mit reverser Transkriptase (RT-PCA) entwickelt. Für eine schnelle und einfache Handhabung wurde auch dieser Assay von Beginn an so konzipiert und optimiert, dass ein Nachweis ohne vorherige RNA-Extraktion direkt aus den Tupfern erfolgen kann.
Ergebnisse
Yersinia Pestis
Mit dem im Rahmen des Projektes entwickelten pla-spezifischen Assay gelang der Nachweis von Yersinia pestis zuverlässig und innerhalb von nur 20 min sowohl aus Flüssigkulturmaterial als auch aus Sputumproben (Abbildung 2A). Das mittels Probit-Analyse festgestellte Detektionslimit (KI 95%) lag dabei bei 35 Bakterien (Kulturmaterial) bzw. 48 Bakterien (Sputum) pro Reaktion und lag damit deutlich unter dem Detektionslimit der klassischen Schnelltests. Gleichzeitig wurden keinerlei Kreuzreaktivitäten mit nah- und fernverwandten Bakterien festgestellt, sodass sich der Assay als vollkommen spezifisch für Yersinia pestis erwies. Durch ein initiales 5-minütiges Erhitzen (66°C) der unbehandelten Proben vor der eigentlichen Amplifikation konnte auf eine DNA-Extraktion verzichtet werden. Die Amplifikation fand im Anschluss mit 550 Zyklen je 1,5 s (Gesamtlaufzeit: 13,75 min) statt (Abbildung 2). Durch die Nutzung des gefriergetrockneten Mastermixes, der lediglich in 40µl Wasser gelöst werden muss, konnte der Arbeitsaufwand auf ein Minimum reduziert und eine Durchführung selbst im schweren Schutzanzug ermöglicht werden. Dies konnte im Rahmen der Übung „Precise Response“ unter realen Bedingungen erfolgreich unter Beweis gestellt werden (Abbildung 2B).
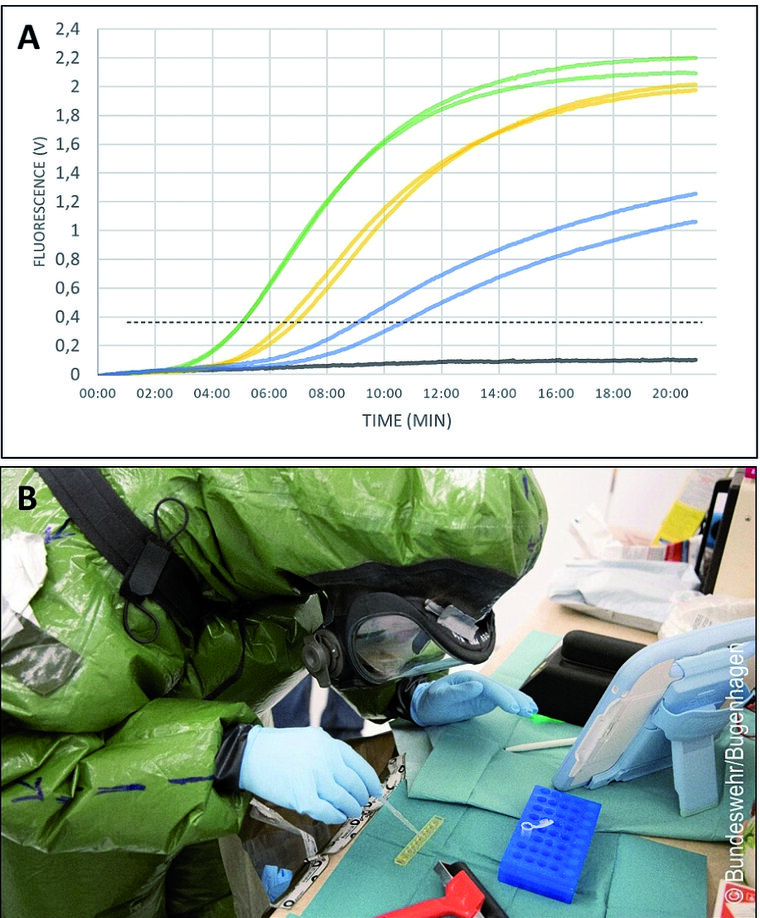
Abb. 2: (A) Nachweis von Yersinia pestis aus Sputumproben (jeweils im Doppelansatz); (B) Anwendung im Feld im Rahmen eines simulierten RDOIT Einsatzes
SARS-CoV-2
SARS-CoV-2 konnte mittels RT-PCA hochspezifisch und ohne vorherige Nukleinsäureextraktion direkt aus Abstrichtupfern nachgewiesen werden. Das Detektionslimit (KI 95%) der Probit-Analyse lag dabei bei nur 4,9 Kopien pro Mikroliter. In Abhängigkeit von der ursprünglichen RNA-Menge in der Probe dauerte ein positiver Nachweis zwischen fünf und 24 Minuten und ist damit der herkömmlichen PCR zeitlich deutlich überlegen. Insgesamt wurden 83 SARS-CoV-2-positive Patientenproben untersucht, wovon 89,2% (n = 74) erfolgreich mittels PCA nachgewiesen werden konnten. Dabei konnten 100% der Proben, die in der herkömmlichen PCR einen ct-Wert von ≤ 25 aufwiesen, erfolgreich detektiert werden.
Diskussion und Schlussfolgerung
Die PCA ist eine neue Methode zur Erregeridentifikation mittels Nukleinsäurenachweis. Sie beruht auf dem Prinzip der klassischen PCR, ist jedoch mit deutlich weniger Material- und Zeitaufwand verbunden. Im Rahmen des Projektes konnten zwei diagnostische Assays zum Nachweis von Yersinia pestis bzw. SARS-CoV-2 entwickelt und evaluiert werden.
Dank des nukleinsäurebasierten Nachweises ist die Yersinia pestis-PCA den klassischen Schnelltests sowohl in Sensitivität als auch in Spezifität deutlich überlegen. Im direkten Vergleich mit dem Goldstandard ist der von uns entwickelte Assay der PCR im Hinblick auf Sensitivität unterlegen während sich die Spezifität nicht unterscheidet. Hier könnte unter Umständen eine Optimierung der Zielsequenzen zu einer Verbesserung der Empfindlichkeit führen. Denkbar wären hier vor allem der Einsatz von multi-copy Targets wie beispielsweise 16S oder 23S ribosomale RNA. Obwohl wir bei der Evaluation des Yersinia pestis Assays keinerlei Kreuzreaktivität feststellen konnten, muss beachtet werden, dass das pla-Gen in der Literatur als nicht 100% spezifisch für Yersinia pestis beschrieben wurde. Hier könnte die Entwicklung eines multiplex Assays mit einem zweiten Target (z. B. das caf1-Gen [2]) in Erwägung gezogen werden, um mehr Sicherheit zu schaffen.
Mithilfe des von uns entwickelten SARS-CoV-2 Assays ist es möglich, SARS-CoV-2-Infektionen innerhalb kürzester Zeit direkt aus Abstrichtupfern nachzuweisen. In Abhängigkeit von der Virusmenge in der Probe kann ein positives Ergebnis teilweise bereits nach fünf Minuten abgelesen werden, womit der Assay der klassischen PCR deutlich überlegen ist. In Bezug auf die Sensitivität erreicht auch der SARS-CoV-2 Assay nicht ganz die Empfindlichkeit einer herkömmlichen PCR. Jedoch liegt das ermittelte Detektionslimit deutlich unter der durchschnittlichen Viruslast, die für Patienten aller Erkrankungsgrade beschrieben wurde [3]. Basierend darauf kann davon ausgegangen werden, dass der SARS-CoV-2-Assay in der Lage ist, die große Mehrheit an Infektionen in klinischen Proben nachzuweisen. In jedem Fall ist er in der Lage, eine Infektion bei den klinisch besonders relevanten hochkontagiösen Patienten zu diagnostizieren. Durch die einfache Handhabung sowie die kurzen Laufzeiten kann die PCA dabei völlig laborunabhängig beispielsweise direkt beim Patienten eingesetzt werden.
Zusammenfassend konnte gezeigt werden, dass die Erregeridentifizierung mittels PCA funktioniert und sich sowohl für die Nutzung als sichere und schnelle Point-of-Care Diagnostik als auch im Feld einsetzen lässt. Sie ist damit eine wichtige Erweiterung des Methodenspektrums des medizinischen B-Schutzes.
Literatur
- Corman VM, Landt O, Kaiser M et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance 2020; 25(3): 2000045. mehr lesen
- Engelthaler DM, Gage KL, Montenieri JA, Chu M, Carter LG: PCR Detection of Yersinia pestis in Fleas: Comparison with Mouse Inoculation. J Clin Microbiol 1999; 37(6): 1980–1984. mehr lesen
- Jones TC, Biele G, Mühlemann B et al.: Estimating infectiousness throughout SARS-CoV-2 infection course. Science 2021: 373(6551): eabi5273. mehr lesen
- Parkhill J, Wren BW, Thomson NR et al.: Genome sequence of Yersinia pestis, the causative agent of plague. Nature 2001; 13(6855): 523–527. mehr lesen
- Riehm JM, Rahalison L, Scholz HC et al.: Detection of Yersinia pestis using real-time PCR in patients with suspected bubonic plague. Mol Cell Probes 2011; 25(1): 8–12. mehr lesen
- Ziegler I, Vollmar P, Knüpfer M, Braun P, Stoecker K: Reevaluating limits of detection of 12 lateral flow immunoassays for the detection of Yersinia pestis, Francisella tularensis, and Bacillus anthracis spores using viable risk group-3 strains. J Appl Microbiol 2021: 130: 1173-1180. mehr lesen
Manuskriptdaten
Zitierweise
Müller K, Zwirglmaier K, Daßen S, Stehr J, Ullerich L, Stoecker K: Pulse-Controlled Amplification – eine neuartige Methode für die Erregeridentifizierung mittels Nukleinsäurenachweis (Vortrags-Abstract). WMM 2021; 65(11): S14-S16..
Für die Verfasser
Oberstabsveterinär Dr. Katharina Müller
Institut für Mikrobiologie der Bundeswehr
Neuherbergstr. 11, 80937 München
E-Mail: katharina5mueller@bundeswehr.org
Vortrag beim Wettbewerb um den Heinz-Gerngroß-Förderpreis der Deutschen Gesellschaft für Wehrmedizin und Wehrpharmazie e. V. am 15. Oktober 2021 in Koblenz